目录
Heterogeneous Effect over Time
Heterogeneous Effect over Time
有好消息也有坏消息。首先是好消息:你已经发现了问题所在。也就是说,你知道 TWFE 在应用于具有时间异构效应的交错采用数据时是有偏差的。用更专业的术语来说,您的数据生成过程具有不同的效应参数:
但你假设效果是恒定的:
如果问题出在这里,一个简单的解决方法就是让每个时间和单位都有不同的效果。理论上,你可以用下面的公式实现类似的效果:downloads ~ treated:post:C(date):C(city) + C(date) + C(city)
就这样?问题解决了?不尽然。坏消息是:这个模型的参数会多于数据点的数量。由于日期和单位是相互影响的,因此每个单位在每个时间段都会有一个干预效应参数。但这正是您所拥有的样本数量!OLS 在这里根本无法运行。
好吧,所以你需要减少模型的干预效应参数数量。要做到这一点,你能想到以某种方式对单位进行分组吗?如果你稍微挠挠头,就会发现一种非常自然的分组方法:按队列分组!要知道,整个组群的效应随着时间的推移会遵循相同的模式。因此,对这种不切实际的模式的一个自然改进就是允许效应按群组而不是按单位变化:
该模型的干预效果参数数量更合理,因为队列数通常比单位数少得多。现在,您终于可以运行模型了:
formula = "downloads ~ treated:post:C(cohort):C(date) + C(city)+C(date)"
twfe_model = smf.ols(formula, data=mkt_data_cohorts_w).fit()这将为您提供多个 ATT 估计值:每个队列和日期一个。因此,要想知道自己的方法是否正确,可以计算模型所隐含的个体干预效果估计值,然后求出平均值。要做到这一点,只需将干预后各单位的实际干预结果与模型预测的 进行比较即可:
df_pred = (
mkt_data_cohorts_w
.query("post==1 & treated==1")
.assign(y_hat_0=lambda d: twfe_model.predict(d.assign(treated=0)))
.assign(effect_hat=lambda d: d["downloads"] - d["y_hat_0"])
)
print("Number of param.:", len(twfe_model.params))
print("True Effect: ", df_pred["tau"].mean())
print("Pred. Effect: ", df_pred["effect_hat"].mean())
Number of param.: 510
True Effect: 2.2625252108176266
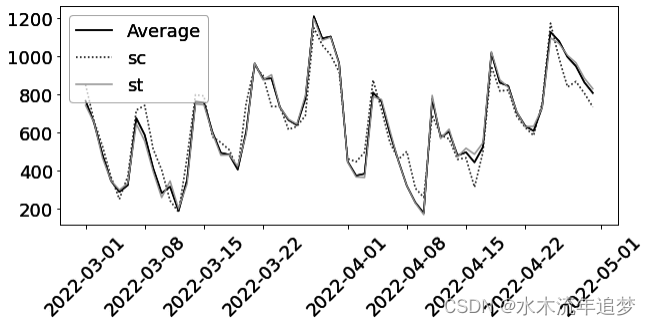
Pred. Effect: 2.259766144685074最终这给了你一个带有一堆参数的模型,但它确实设法恢复了真正的ATT。你甚至可以提取这些att并绘制它们:
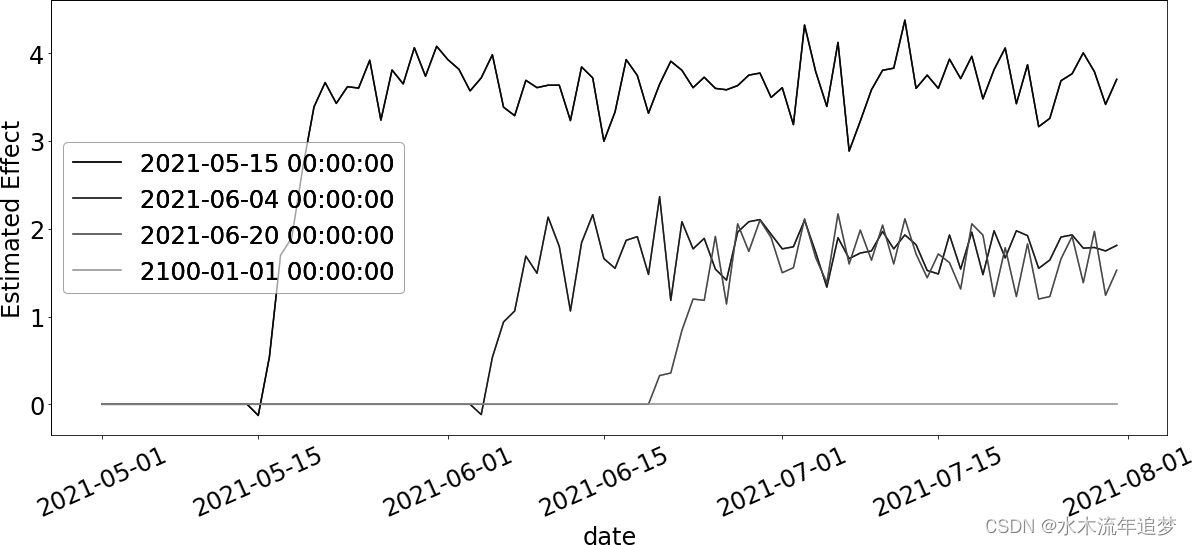
这幅图的好处在于,它符合您对效应表现的直觉:效应逐渐上升,一段时间后保持不变。此外,它还告诉您,在干预前的所有时期,效果都为零,因此,从未干预过的人群的效果也为零。这可能会给您提供一些如何减少该模型参数数量的思路。例如,您可以只考虑大于队列的时间段的影响:
这将涉及大量的特性工程,因为你必须在治疗前对日期进行分组,但很高兴知道这是可能的。
就像在差异模型中加入协变量的问题一样,解决 TWFE 偏差的方法有两种。一种是在运行双向固定效应模型时巧妙地将虚拟变量进行交互。另一种方法是将问题分解成多个 2 × 2 差分模型,分别求解每个模型,然后合并结果。一种方法是使用从未接受过治疗的组作为对照,为每个队列估计一个差异模型:
cohorts = sorted(mkt_data_cohorts_w["cohort"].unique())
treated_G = cohorts[:-1]
nvr_treated = cohorts[-1]
def did_g_vs_nvr_treated(df: pd.DataFrame,
cohort: str,
nvr_treated: str,
cohort_col: str = "cohort",
date_col: str = "date",
y_col: str = "downloads"):
did_g = (
df
.loc[lambda d:(d[cohort_col] == cohort)|
(d[cohort_col] == nvr_treated)]
.assign(treated = lambda d: (d[cohort_col] == cohort)*1)
.assign(post = lambda d:(pd.to_datetime(d[date_col])>=cohort)*1)
)
att_g = smf.ols(f"{y_col} ~ treated*post",
data=did_g).fit().params["treated:post"]
size = len(did_g.query("treated==1 & post==1"))
return {"att_g": att_g, "size": size}
atts = pd.DataFrame(
[did_g_vs_nvr_treated(mkt_data_cohorts_w, cohort, nvr_treated)
for cohort in treated_G]
)
atts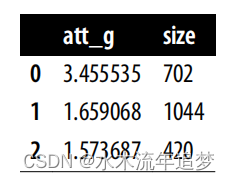
然后,您可以将结果与加权平均值相结合,其中的权重是每个队列的样本量(T * N)。得出的估计结果与您之前的估计结果非常相似:
(atts["att_g"]*atts["size"]).sum()/atts["size"].sum()
2.2247467740558697另外,您也可以使用尚未接受治疗的人群作为对照,而不是使用从未接受治疗的人群作为对照,这样可以增加对照的样本量。这种方法比较麻烦,因为您必须对同一队列多次运行差分法。