简介
DeepSequence 是一个生成性的、无监督的生物序列潜变量模型。给定一个多重序列比对作为输入,它可以用来预测可获得的突变,提取监督式学习的定量特征,并生成满足明显约束的新序列文库。它将序列中的高阶依赖性建模为残差子集之间约束的非线性组合。要了解更多信息,请查看论文(https://www.biorxiv.org/content/early/2017/12/18/235655.1)和下面的例子。
为了便于分析,我们建议使用 EVcouplings package(https://github.com/debbiemarkslab/EVcouplings)程序包生成对齐,尽管可以使用任何序列比对。
例子
对于合理的培训时间,我们建议在 GPU 上培训 DeepSequence:
THEANO_FLAGS='floatX=float32,device=cuda' python run_svi.py
但是,它可以在 CPU 上运行:
python run_svi.py
示例子文件夹中的 iPython 笔记本中提供了该分析的其他用法示例和特性。
注:.ipynb结尾的文件格式需要用jupyter notebook打开。
例子1-1 下载例子文件中需要的数据(shell中运行)
bash download_alignments.sh
bash download_pretrained.sh
注:这里有个bug,下载后的aligments文件放在了example下的aligments文件夹下,后面发现例子文件中给出的调用代码中使用的地址是datasets文件夹下,这里确实是例子文件中的调用地址写错了,我是将这个aligments文件夹名更改成了datasets,后续才能正常运行。
例子1-2 探索模型的输出
import theano
import numpy as np
import sys
%matplotlib inline
import matplotlib.pyplot as plt
注:我在运行%matplotlib inline时报错
>>> %matplotlib inline
File "<stdin>", line 1
%matplotlib inline
^
SyntaxError: invalid syntax
这是因为我是直接使用的linux系统中的原生python,而该命令属于jupyter notebook的中魔法命令,修改后的命令如下
import theano
import numpy as np
import sys
import matplotlib
matplotlib.use('TkAgg')
#%matplotlib inline
import matplotlib.pyplot as plt
例子1-3 加载DeepSequence软件的本地模块
sys.path.insert(0, "../DeepSequence")
import model
import helper
import train
例子1-4 构建模型
data_params = {
"dataset":"BLAT_ECOLX"}
data_helper = helper.DataHelper(
dataset=data_params["dataset"],
working_dir=".",
calc_weights=False
)
model_params = {
"batch_size" : 100,
"encode_dim_zero" : 1500,
"encode_dim_one" : 1500,
"decode_dim_zero" : 100,
"decode_dim_one" : 500,
"n_patterns" : 4,
"n_latent" : 30,
"logit_p" : 0.001,
"sparsity" : "logit",
"encode_nonlin" : "relu",
"decode_nonlin" : "relu",
"final_decode_nonlin": "sigmoid",
"output_bias" : True,
"final_pwm_scale" : True,
"conv_pat" : True,
"d_c_size" : 40
}
vae_model = model.VariationalAutoencoder(data_helper,
batch_size = model_params["batch_size"],
encoder_architecture = [model_params["encode_dim_zero"],
model_params["encode_dim_one"]],
decoder_architecture = [model_params["decode_dim_zero"],
model_params["decode_dim_one"]],
n_latent = model_params["n_latent"],
n_patterns = model_params["n_patterns"],
convolve_patterns = model_params["conv_pat"],
conv_decoder_size = model_params["d_c_size"],
logit_p = model_params["logit_p"],
sparsity = model_params["sparsity"],
encode_nonlinearity_type = model_params["encode_nonlin"],
decode_nonlinearity_type = model_params["decode_nonlin"],
final_decode_nonlinearity = model_params["final_decode_nonlin"],
output_bias = model_params["output_bias"],
final_pwm_scale = model_params["final_pwm_scale"],
working_dir = ".")
print ("Model built")
显示结果
Encoding sequences
Neff = 8355.0
Data Shape = (8355, 253, 20)
Model built
注:由于使用的原生python解释器,这里每一部分我都是分开运行的。
例子1-5 构建模型(装载训练好的参数)
file_prefix = "BLAT_ECOLX"
vae_model.load_parameters(file_prefix=file_prefix)
print ("Parameters loaded")
结果
Parameters loaded
例子1-5 打印目标序列的信息
打印
print (data_helper.focus_seq_name)
print (str(data_helper.focus_start_loc)+"-"+str(data_helper.focus_stop_loc))
print (data_helper.focus_seq)
结果
>BLAT_ECOLX/24-286
24-286
hpetlVKVKDAEDQLGARVGYIELDLNSGKILeSFRPEERFPMMSTFKVLLCGAVLSRVDAGQEQLGRRIHYSQNDLVEYSPVTEKHLTDGMTVRELCSAAITMSDNTAANLLLTTIGGPKELTAFLHNMGDHVTRLDRWEPELNEAIPNDERDTTMPAAMATTLRKLLTGELLTLASRQQLIDWMEADKVAGPLLRSALPAGWFIADKSGAGErGSRGIIAALGPDGKPSRIVVIYTTGSQATMDERNRQIAEIGASLIkhw
例子1-6 去除小写字母
print ("".join(data_helper.focus_seq_trimmed))
结果
VKVKDAEDQLGARVGYIELDLNSGKILSFRPEERFPMMSTFKVLLCGAVLSRVDAGQEQLGRRIHYSQNDLVEYSPVTEKHLTDGMTVRELCSAAITMSDNTAANLLLTTIGGPKELTAFLHNMGDHVTRLDRWEPELNEAIPNDERDTTMPAAMATTLRKLLTGELLTLASRQQLIDWMEADKVAGPLLRSALPAGWFIADKSGAGEGSRGIIAALGPDGKPSRIVVIYTTGSQATMDERNRQIAEIGASLI
例子1-7 求一下模型序列的潜在变量
focus_seq_one_hot = np.expand_dims(data_helper.one_hot_3D(data_helper.focus_seq_trimmed),axis=0)
mu_blat, log_sigma_blat = vae_model.recognize(focus_seq_one_hot)
print ("mu:")
print (mu_blat[0])
print ("\nlog sigma:")
print (log_sigma_blat[0])
结果
mu:
[-2.93129622e-02 2.80484591e-02 4.74045508e-02 2.08589889e-02
-1.58986675e-02 -1.26072732e+00 -1.66292705e-02 1.40488074e+00
1.67025877e-02 -8.53566889e-03 2.18774280e-02 2.32125783e+00
2.55409795e-02 -1.15651891e-02 -3.02552657e-02 1.24139726e-02
-4.12962164e-02 -8.41912007e-01 -5.99993306e-03 1.61336532e-03
6.03782405e-01 3.46253082e-02 2.27598501e-02 -2.05355599e-02
3.94717697e-03 1.35123502e+00 8.65776037e-03 -7.57649777e-03
3.81364257e-02 -2.08573404e-02]
log sigma:
[-4.22987289e-04 -1.84314600e-02 -4.19494449e-02 -3.17123838e-02
-1.67881093e-02 -2.28357830e+00 -5.77861108e-02 -9.92522460e-01
-3.17816964e-02 -1.74817094e-02 -4.95498244e-02 -1.99601526e+00
-2.00790022e-02 2.04198661e-02 -1.36289102e-02 -6.93278173e-03
-2.53706808e-02 -1.90622283e+00 -2.70597848e-02 -1.67551476e-02
-2.16649542e+00 1.02254690e-02 8.96915176e-03 -1.85787921e-02
2.56248426e-02 -2.42973459e+00 -3.70118837e-02 1.25962615e-02
2.43832070e-02 -6.73871211e-03]
例子1-8 从中取样
z_blat = vae_model.encode(focus_seq_one_hot)
print ("z:")
print (z_blat[0])
结果
z:
[ 1.226298 -0.50005774 -0.05264941 0.66986938 -0.71224884 -1.15921493
0.05177535 1.1583736 0.92446647 -0.14730169 0.78172059 2.35628846
1.32076451 1.1621947 -0.12593087 -0.49765921 0.55820479 -0.84410041
-1.94164654 1.19722414 0.51352968 0.23148123 0.56776408 -0.36881278
-0.590448 1.50986844 0.68191917 -1.4255378 -0.08560662 1.08017904]
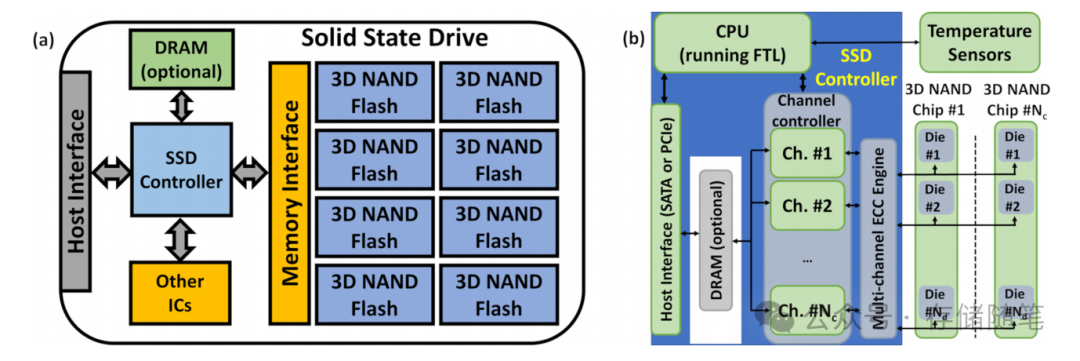
例子1-9 然后通过采样的潜在变量生成序列周围的样本分布
seq_reconstruct = vae_model.decode(z_blat)
plt.figure(figsize=(35,10))
plt.imshow(seq_reconstruct[0].T,cmap=plt.get_cmap("Blues"))
ax = plt.gca()
ax.set_yticks(np.arange(len(data_helper.alphabet)))
ax.set_yticklabels(list(data_helper.alphabet))
plt.show()
结果


















![[.NET] 查询当前已安装所有 Win32 与 UWP 应用](https://img-blog.csdnimg.cn/img_convert/8bc34826fea544454d22dd06e84931ce.png)






![PyTorch][chapter 13[李宏毅深度学习][Semi-supervised Linear Methods-2]](https://img-blog.csdnimg.cn/direct/043cd316533c47919465de78ade47590.png)