介绍
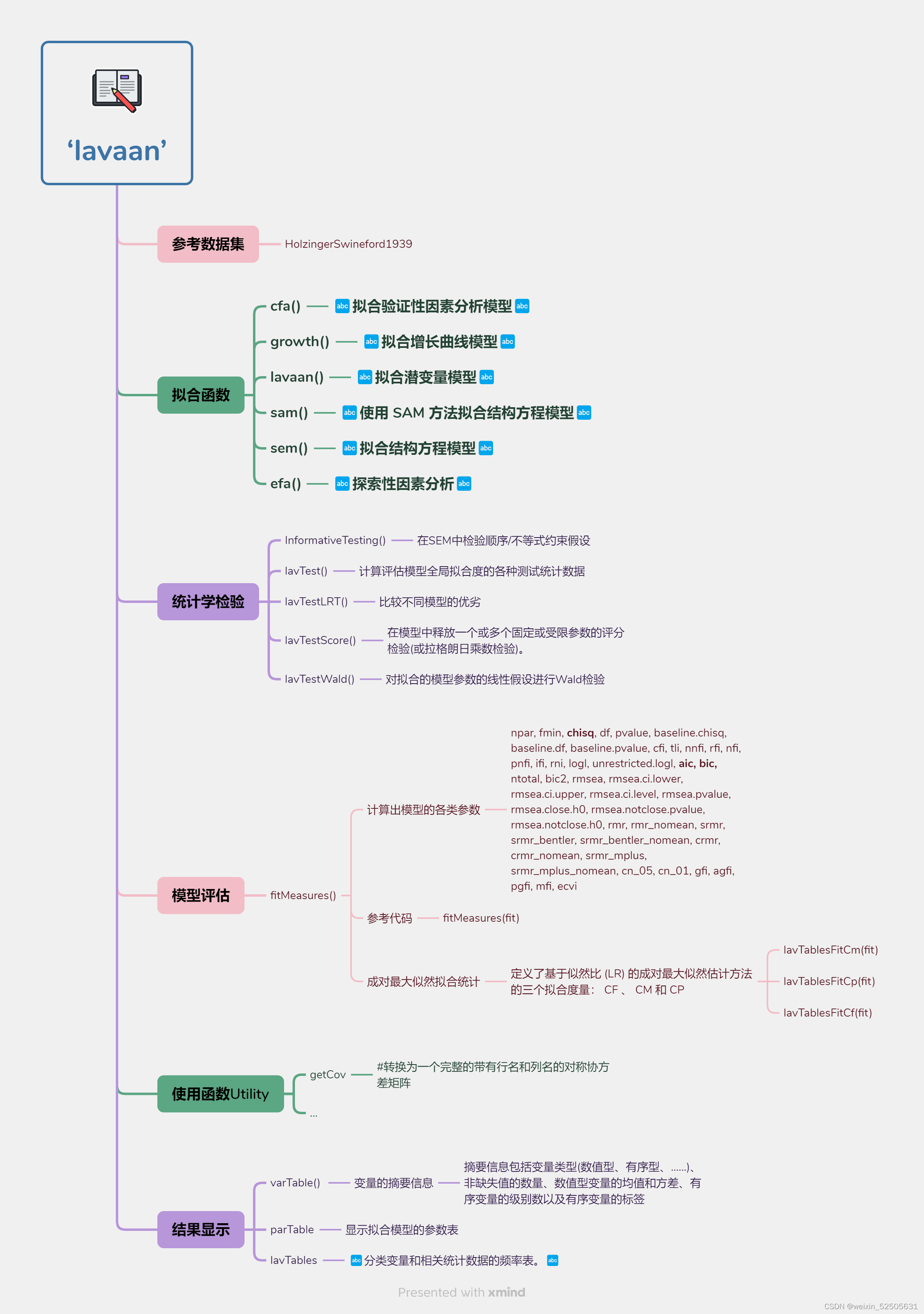
PTXQC包是2016年发表在J Proteome Res期刊上的R包,它主要是对MaxQuant输出结果进行提取处理从而获得评估蛋白质质量结果。
安装
从github安装,安装过程会自动构建tutorial。
devtools::install_github("cbielow/PTXQC", build_vignettes = TRUE, dependencies = TRUE)
library(PTXQC)
- 查看帮助文档,帮助文档是以html方式展示
help(package="PTXQC")
browseVignettes(package = 'PTXQC')

输入文件
输入文件是MaxQuant结果文件的txt里面的:/combined/txt
- parameters.txt
- summary.txt
- proteinGroups.txt
- evidence.txt
- msms.txt
- msmsScans.txt
运行
输入包含上述输入文件的目录即可,然后使用createReport函数。
r = createReport(txt_folder)
cat(paste0("\nReport generated as '", r$report_file, "'\n\n"))
它也提供了修改报告主题或者评估步骤选择的方法,需要修改yaml_file文件,可参考如下。
require(PTXQC)
require(yaml)
## the next require() is needed to prevent a spurious error in certain R versions (might be a bug in R or a package)
## error message is:
## Error in Scales$new : could not find function "loadMethod"
require(methods)
## specify a path to a MaxQuant txt folder
## Note: This folder can be incomplete, depending on your YAML config
if (1) {
## we will use an example dataset from PRIDE (dataset 2 of the PTXQC publication)
local_zip = tempfile(fileext=".zip")
download.file("ftp://ftp.pride.ebi.ac.uk/pride/data/archive/2015/11/PXD003133/txt_20min.zip", destfile = local_zip)
unzip(local_zip, exdir = tempdir()) ## extracts content
txt_folder = file.path(tempdir(), "txt_20min")
} else {
## if you have local MaxQuant output, just use it
txt_folder = "c:/Proteomics/MouseLiver/combined/txt"
}
## use a YAML config inside the target directory if present
fh_out = getReportFilenames(txt_folder)
if (file.exists(fh_out$yaml_file))
{
cat("\nUsing YAML config already present in target directory ...\n")
yaml_config = yaml.load_file(input = fh_out$yaml_file)
} else {
cat("\nYAML config not found in folder '", txt_folder, "'. The first run of PTXQC will create one for you.", sep="")
yaml_config = list()
}
r = createReport(txt_folder, mztab_file = NULL, yaml_obj = yaml_config)
cat(paste0("\nReport generated as '", r$report_file, "'\n\n"))
结果
输出的报告文件可以是html也可以是PDF格式,如图:基于PTXQC包评估以下部分
- 样本制备(1-5);
- 液相色谱分离肽段(6-9);
- 质谱过程(10-18);
- 鉴定蛋白效果(19-22)。

不同颜色代表实验过程的优劣。从图中可以看出,样本制备和质谱过程获得评分是best,而鉴定蛋白质效果这一步效果最差,这是因为我们的蛋白质样本是血液外泌体蛋白质,它含量相对血液而已本身就较少,无法达到该包给的阈值(Peptide Count > 15,000; Protein Count > 3,500),但我们可以看到Average Overall Quality是偏较好评价的(偏深绿色)。另外我们蛋白质质谱过程使用了PAIMS技术分离蛋白质,这也是我们能看到每个file有三个bar图(40;60;80电压)。
在获取图谱过程中,常会用到2018年才推出的FAIMS(High-Field Asymmetric Waveform Ion Mobility Spectrometry)技术以用于加载不同电压(肽段在ESI离子化后,进入质谱之前实现快速气相分离,提高分离的峰容量),直接使用多电压下的raw data做MaxQuant定量分析是错误的,MaxQuant软件只能识别单电压的raw data,因此需要使用FAIMS MzXML Generator 软件将raw data转换成各自电压下的MzXML文件。
List of metrics

systemic information
sessionInfo()
R version 4.0.2 (2020-06-22)
Platform: x86_64-conda_cos6-linux-gnu (64-bit)
Running under: CentOS Linux 8 (Core)
Matrix products: default
BLAS/LAPACK: /disk/share/anaconda3/lib/libopenblasp-r0.3.10.so
locale:
[1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8
[5] LC_MONETARY=en_US.UTF-8 LC_MESSAGES=en_US.UTF-8 LC_PAPER=en_US.UTF-8 LC_NAME=C
[9] LC_ADDRESS=C LC_TELEPHONE=C LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] PTXQC_1.0.12 tibble_3.1.5 dplyr_1.0.7
loaded via a namespace (and not attached):
[1] tinytex_0.32 tidyselect_1.1.1 xfun_0.24 bslib_0.2.5.1 reshape2_1.4.4 purrr_0.3.4
[7] colorspace_2.0-2 vctrs_0.3.8 generics_0.1.0 viridisLite_0.4.0 htmltools_0.5.1.1 yaml_2.2.1
[13] utf8_1.2.1 rlang_0.4.11 jquerylib_0.1.4 pillar_1.6.4 glue_1.4.2 DBI_1.1.1
[19] gdtools_0.2.2 RColorBrewer_1.1-2 lifecycle_1.0.0 plyr_1.8.6 stringr_1.4.0 munsell_0.5.0
[25] gtable_0.3.0 rvest_0.3.6 kableExtra_1.3.4 evaluate_0.14 knitr_1.33 UpSetR_1.4.0
[31] fansi_0.5.0 Rcpp_1.0.7 scales_1.1.1 webshot_0.5.2 jsonlite_1.7.2 systemfonts_0.3.2
[37] gridExtra_2.3 ggplot2_3.3.5 digest_0.6.27 stringi_1.4.6 ade4_1.7-18 cowplot_1.1.0
[43] grid_4.0.2 tools_4.0.2 magrittr_2.0.1 sass_0.4.0 ggdendro_0.1.22 R6P_0.2.2
[49] seqinr_4.2-4 crayon_1.4.1 tidyr_1.1.4 pkgconfig_2.0.3 ellipsis_0.3.2 MASS_7.3-54
[55] data.table_1.14.0 xml2_1.3.2 assertthat_0.2.1 rmarkdown_2.9 svglite_1.2.3.2 httr_1.4.2
[61] rstudioapi_0.13 R6_2.5.0 compiler_4.0.2
Reference
Proteomics quality control: quality control software for MaxQuant results