DeepSORT(目标跟踪算法)中的数值表格与调参的关系
flyfish
代码地址
https://github.com/shaoshengsong/DeepSORT
文字要是懒得看,直接拖到后面看图。
原始代码
Python版
chi2inv95 = {
1: 3.8415,
2: 5.9915,
3: 7.8147,
4: 9.4877,
5: 11.070,
6: 12.592,
7: 14.067,
8: 15.507,
9: 16.919}
C++版
const double KalmanFilter::chi2inv95[10] = {
0,
3.8415,
5.9915,
7.8147,
9.4877,
11.070,
12.592,
14.067,
15.507,
16.919};
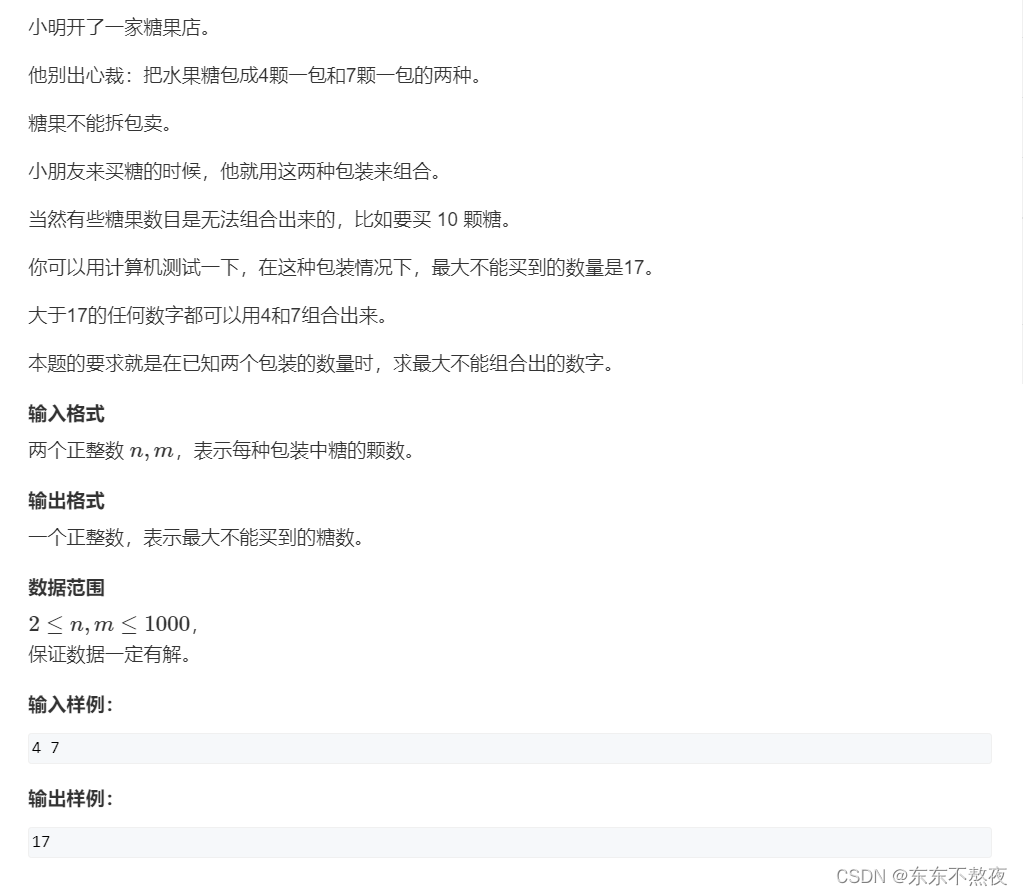
原始的这些数值表格
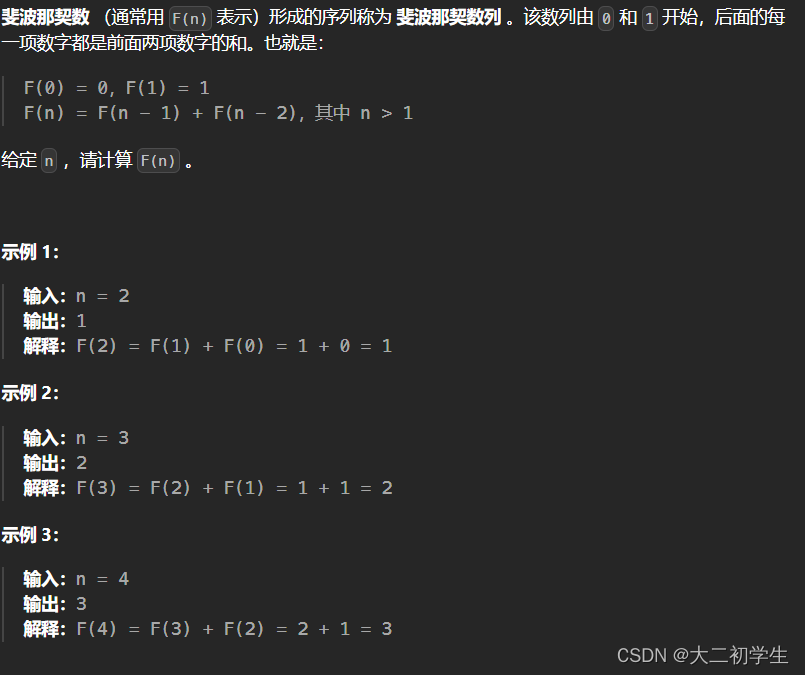
表示卡方分布在自由度为N时的0.95分位数。这些数值是通过统计软件(如MATLAB或Octave)的chi2inv函数计算出来的。chi2inv函数计算的是卡方分布的逆累积分布函数(inverse cumulative distribution function),即给定一个概率(这里是0.95),求对应的卡方分布的值。这些数值用于马氏距离的阈值。卡方分布(chi-square distribution)是用于统计学中的一种概率分布,通常用于假设检验和方差分析。对于给定的自由度N,卡方分布的0.95分位数表示在该自由度下,随机变量的值有95%的概率落在此分位数之下。
自己实现一个
数值计算方法:
假设我们使用Python和SciPy库来计算这些值,可以通过以下代码实现:
import scipy.stats as stats
# 定义自由度列表
degrees_of_freedom = range(1, 10)
# 计算每个自由度下的0.95分位数
chi2inv95 = {df: stats.chi2.ppf(0.95, df) for df in degrees_of_freedom}
print(chi2inv95)
解释计算结果:
- 自由度1: 对应的0.95分位数是3.8415,这意味着在自由度为1的情况下,随机变量有95%的概率其值在3.8415以下。
- 自由度2: 对应的0.95分位数是5.9915,这意味着在自由度为2的情况下,随机变量有95%的概率其值在5.9915以下。
依此类推,直至自由度为9。
使用场景:
在DeepSort算法中,马氏距离用于衡量目标检测之间的相似性。卡方分布的0.95分位数作为阈值,是为了确保在给定的置信水平下,可以有效地过滤掉不相似的目标检测,从而提高跟踪的准确性。换句话说,如果两个检测的马氏距离大于对应自由度的0.95分位数,那么它们被认为是不同的目标。
输出
{1: 3.841458820694124,
2: 5.991464547107979,
3: 7.814727903251179,
4: 9.487729036781154,
5: 11.070497693516351,
6: 12.591587243743977,
7: 14.067140449340167,
8: 15.507313055865453,
9: 16.918977604620448}
马氏距离
马氏距离(Mahalanobis Distance)是一种度量点与点之间距离的方法,考虑了数据的相关性和尺度。具体来说,马氏距离计算两个点之间的距离时,使用了数据的协方差矩阵,使得它在数据的不同维度上有不同的缩放,能够更准确地反映点与点之间的关系。
马氏距离的公式如下:
D M ( x , y ) = ( x − y ) T S − 1 ( x − y ) D_M(\mathbf{x}, \mathbf{y}) = \sqrt{(\mathbf{x} - \mathbf{y})^T \mathbf{S}^{-1} (\mathbf{x} - \mathbf{y})} DM(x,y)=(x−y)TS−1(x−y)
其中:
- x \mathbf{x} x 和 y \mathbf{y} y 是两个数据点的向量。
- S \mathbf{S} S 是协方差矩阵。
- S − 1 \mathbf{S}^{-1} S−1 是协方差矩阵的逆矩阵。
马氏距离用于测量两个点之间的多维空间距离,其考虑了数据的协方差结构。阈值是在特定的置信水平下,用于判断两个点是否属于同一类别的界限。在DeepSort算法中,马氏距离的阈值对应于卡方分布的0.95分位数。如果两个检测的马氏距离超过了这个阈值,则认为它们不属于同一个目标。
动画展示
展示了马氏距离随给定点变化的情况,不同位置的给定点与数据集中其他点的马氏距离
(播放一遍就停了,没有循环播,需要刷新一下再看)
pip install matplotlib seaborn numpy scipy
pip install imageio
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from scipy.spatial.distance import mahalanobis
from scipy.stats import chi2
import imageio
# 生成示例数据
np.random.seed(42)
mean = [0, 0]
cov = [[1, 0.5], [0.5, 1]]
data = np.random.multivariate_normal(mean, cov, 500)
# 计算马氏距离的函数
def calculate_mahalanobis_distance(data, point):
cov_matrix = np.cov(data, rowvar=False)
inv_cov_matrix = np.linalg.inv(cov_matrix)
mean_data = np.mean(data, axis=0)
distances = [mahalanobis(d, point, inv_cov_matrix) for d in data]
return distances
# 绘制散点图并添加马氏距离
def plot_mahalanobis(data, point, frame_number):
plt.figure(figsize=(10, 8))
sns.scatterplot(x=data[:, 0], y=data[:, 1], hue=calculate_mahalanobis_distance(data, point), palette='viridis')
plt.scatter(point[0], point[1], color='red')
plt.colorbar(label='Mahalanobis Distance')
plt.title(f'Mahalanobis Distance at Frame {frame_number}')
plt.xlabel('X')
plt.ylabel('Y')
plt.savefig(f'frame_{frame_number}.png')
plt.close()
# 定义动画点
points = np.linspace(-3, 3, 30)
frames = []
# 生成每一帧图像
for i, p in enumerate(points):
plot_mahalanobis(data, [p, p], i)
frames.append(imageio.imread(f'frame_{i}.png'))
# 保存为GIF
imageio.mimsave('mahalanobis_distance.gif', frames, fps=2)

生成示例数据:使用 np.random.multivariate_normal 生成500个二维数据点,服从均值为 [0, 0] 和协方差矩阵为 [[1, 0.5], [0.5, 1]] 的多元正态分布。
计算马氏距离的函数:calculate_mahalanobis_distance 函数计算数据集中每个点与给定点的马氏距离。
绘制散点图并添加马氏距离:plot_mahalanobis 函数绘制散点图,并根据马氏距离着色。给定点用红色标出。
定义动画点:在 [-3, 3] 区间内生成30个点,作为动画中的给定点。
协方差
协方差(Covariance)是衡量两个变量共同变化程度的统计量。如果两个变量的协方差为正,意味着这两个变量一起增加或减少;如果协方差为负,意味着一个变量增加时另一个变量减少。
协方差的公式如下:
Cov ( X , Y ) = ∑ i = 1 n ( X i − X ˉ ) ( Y i − Y ˉ ) n − 1 \text{Cov}(X, Y) = \frac{\sum_{i=1}^{n} (X_i - \bar{X})(Y_i - \bar{Y})}{n-1} Cov(X,Y)=n−1∑i=1n(Xi−Xˉ)(Yi−Yˉ)
其中:
- X X X 和 Y Y Y 是两个随机变量。
- X i X_i Xi 和 Y i Y_i Yi 是对应的观测值。
- X ˉ \bar{X} Xˉ 和 Y ˉ \bar{Y} Yˉ 是 X X X 和 Y Y Y 的均值。
- n n n 是样本数量。
协方差矩阵是包含所有变量对之间协方差的矩阵,用于多维数据的分析。
自由度(Degrees of Freedom)
自由度(Degrees of Freedom, df)在统计学中,表示用于估算一个统计量的独立信息数量。对于样本方差,自由度等于样本数量减去一个,这是因为我们在计算样本方差时使用了样本均值(这个均值本身也来自样本数据)。因此,我们失去了一个自由度来计算均值,剩下的自由度用于估算方差。
在计算样本方差时,我们首先计算样本均值:
X ˉ = 1 n ∑ i = 1 n X i \bar{X} = \frac{1}{n} \sum_{i=1}^{n} X_i Xˉ=n1i=1∑nXi
样本均值 X ˉ \bar{X} Xˉ 是基于 n n n 个数据点计算出来的,但其实际上只提供了 n − 1 n-1 n−1 个独立信息,因为最后一个数据点的值可以从前 n − 1 n-1 n−1 个数据点的值及均值推断出来。因此,自由度减去1,用以反映我们在估算过程中消耗的一个独立信息。
在统计学中,自由度是指用于计算统计量的独立数值的数量。对于卡方分布,自由度通常对应于所涉及的随机变量的数量。举个简单的例子,如果我们有一个数据集,进行样本方差计算时,自由度等于样本数量减去一个(因为我们要用一个样本均值来估计总体均值)。
0.95分位数
0.95分位数是指在给定分布中,有95%的数据点位于该值以下。对于卡方分布来说,这个值用于判断在95%的置信水平下,观察值是否显著。使用stats.chi2.ppf(0.95, df)可以计算出对应自由度下的0.95分位数。
计算马氏距离并使用卡方分布的0.95分位数作为阈值
import numpy as np
from scipy.stats import chi2
def mahalanobis_distance(x, y, cov):
diff = x - y
inv_cov = np.linalg.inv(cov)
md = np.sqrt(np.dot(np.dot(diff.T, inv_cov), diff))
return md
# 示例数据
x = np.array([1, 2])
y = np.array([1.5, 1.8])
cov = np.array([[1, 0.5], [0.5, 1]])
# 计算马氏距离
md = mahalanobis_distance(x, y, cov)
print(f"Mahalanobis Distance: {md}")
# 设定自由度
degrees_of_freedom = 2
# 计算0.95分位数的卡方门限值
threshold = chi2.ppf(0.95, degrees_of_freedom)
print(f"Chi-square 0.95 quantile for df={degrees_of_freedom}: {threshold}")
# 判断是否匹配
if md < threshold:
print("Match: The points are considered similar.")
else:
print("No Match: The points are considered different.")
计算马氏距离的函数:mahalanobis_distance 计算两个点之间的马氏距离。它接受两个点 x 和 y 以及协方差矩阵 cov 作为输入。
示例数据:定义两个点 x 和 y 及其协方差矩阵 cov。
计算马氏距离:调用 mahalanobis_distance 函数计算两个点之间的马氏距离。
设定自由度:设定自由度为2,因为数据点是二维的。
计算卡方分布的0.95分位数:使用 chi2.ppf 函数计算卡方分布的0.95分位数,作为马氏距离的门限值。
判断是否匹配:通过比较马氏距离与门限值,判断两个点是否匹配。
Mahalanobis Distance: 0.7211102550927978
Chi-square 0.95 quantile for df=2: 5.991464547107979
Match: The points are considered similar.
使用SciPy和Matplotlib来绘制卡方分布的图形,同时标注0.95分位数
import numpy as np
import matplotlib.pyplot as plt
import scipy.stats as stats
# 定义自由度
degrees_of_freedom = [1, 2, 3, 4, 5, 6, 7, 8, 9]
# 设置x轴范围
x = np.linspace(0, 30, 1000)
plt.figure(figsize=(12, 8))
# 绘制不同自由度的卡方分布
for df in degrees_of_freedom:
plt.plot(x, stats.chi2.pdf(x, df), label=f'df={df}')
# 标注0.95分位数
for df in degrees_of_freedom:
chi2_95 = stats.chi2.ppf(0.95, df)
plt.axvline(chi2_95, color='r', linestyle='--')
plt.text(chi2_95, 0.02, f'{chi2_95:.2f}', rotation=90, verticalalignment='bottom')
plt.title('Chi-Square Distribution for Different Degrees of Freedom')
plt.xlabel('Value')
plt.ylabel('Probability Density')
plt.legend()
plt.grid(True)
plt.show()

调参
阈值用于过滤噪声和不相关的目标检测,以提高跟踪算法的准确性和可靠性。在目标跟踪过程中,存在许多不确定性,例如检测误差和环境干扰。通过设置一个合适的阈值,可以在一定的置信水平下排除不可靠的检测结果,从而增强算法的稳健性。具体来说,在DeepSORT算法中,使用卡方分布的0.95分位数作为阈值,可以确保在95%的置信水平下,正确匹配目标检测,从而提高跟踪的准确性。
阈值可以根据应用的需求进行调整。例如,在某些应用中,可以选择更高的置信水平(比如0.99),以获得更严格的匹配标准。只需修改 chi2.ppf 函数中的置信水平即可:
# 计算0.99分位数的卡方门限值
threshold = chi2.ppf(0.99, degrees_of_freedom)
print(f"Chi-square 0.99 quantile for df={degrees_of_freedom}: {threshold}")
这样得到数值再应用到代码中,通过调整置信水平,可以灵活地控制目标匹配的严格程度,从而适应不同的应用场景和需求。















![[ue5]建模场景学习笔记(5)——必修内容可交互的地形,交互沙(3)](https://img-blog.csdnimg.cn/direct/753e06b69d8d4e0f953cc40a706ea906.png)