🤵♂️ 个人主页:@艾派森的个人主页
✍🏻作者简介:Python学习者
🐋 希望大家多多支持,我们一起进步!😄
如果文章对你有帮助的话,
欢迎评论 💬点赞👍🏻 收藏 📂加关注+
喜欢大数据分析项目的小伙伴,希望可以多多支持该系列的其他文章
目录
1.项目背景
我们所经历的全球化进程带来了一系列变化,因为社会、政治、经济和文化进程错综复杂地交织在一起,改变了习俗和习惯,特别是我们的生活方式。这导致与食物过度消费有关的疾病大量增加。肥胖就是一个明显的例子,它越来越常见,因为越来越多的低营养和热量食物被摄入,而且由于交通工具的多种选择以及新的工作和娱乐形式,没有进行必要的体育活动。
世界卫生组织将肥胖和超重描述为在某些身体部位过度积累脂肪,可能对健康有害。自1980年以来,患肥胖症的人数翻了一番,2014年,超过19亿18岁或以上的成年人体重发生变化。超重的一些原因是高脂肪高能量食物的摄入量增加,以及由于久坐工作的性质、新的交通方式和日益城市化而导致的体力活动减少。
肥胖可以被认为是一种具有多种因素的疾病,其症状是体重不受控制地增加,原因是脂肪摄入过多和能量消耗。肥胖可以由遗传背景等生物危险因素引起,因此可以有几种肥胖类型:单基因、瘦素、多基因和综合征。此外,还有其他风险因素,如社会、心理和饮食习惯。另一方面,有人提出了肥胖的其他决定因素,如“独生子女、离婚等家庭冲突、抑郁和焦虑”。
肥胖的发展需要遗传易患这种疾病和暴露于不利环境条件的综合影响。遗传因素决定着组织脂肪形式能量积累的能力或容易程度,而热量形式能量释放的能力或容易程度较低,这意味着肥胖的能源效率较高。产生这种情况的原因是,从长远来看,个人的能源支出低于他所投入的能源,即存在积极的能源平衡。遗传影响与恶劣的饮食习惯和定居生活方式等外部条件有关。
肥胖是一个被称为"世纪流行病"的公共卫生问题,并且根据世界卫生组织的数据表明肥胖的人数持续增加。肥胖这个问题以前与工业化国家有关,但在发展中国家,特别是在城市地区,超重和肥胖现象明显增加。比如,在墨西哥,这是最常见的代谢疾病,作为世界上最肥胖的国家,疟疾流行率从2000年的59.7%上升到2006年的66.7%,对发病率和死亡率居首位的疾病的发展构成了重大风险。成瘾的风险与患糖尿病、高血压、肺和心血管疾病等慢性疾病的高发病率有关,也是发展各种癌症的高风险因素。它还影响到个人的心理领域,降低了受影响者的自尊,影响到他们的社会关系。因此,这一问题的严重程度是显而易见的,而且由于肥胖不分年龄、性别、种族或社会经济地位影响到任何人,情况变得更加令人担忧。
肥胖是一个全球性的公共健康问题,它可以在成人、青少年和儿童中出现。同时,注意到儿童肥胖是成年人肥胖的一个危险因素这一令人震惊的事实,从生命的早期阶段就预防和控制肥胖至关重要,也必须考虑到儿童体重的增加必须是渐进的。由于城市化、经济和技术发展带来的生活方式不断变化,儿童受到影响,导致肥胖儿童人数增加,因此,很多研究集中在对儿童肥胖问题的上。
2.项目简介
2.1项目说明
本文使用UCI中一项关于人们饮食习惯和身体状况调查的数据集,分别通过决策树以及随机森林算法对数据进行处理,拟在寻找肥胖的成因。算法通过对14种影响因子进行多标签分类获取各影响因子与肥胖程度之间的权值,最终获取肥胖评估模型。人们可以通过评估模型就自己目前的生活习惯和身体状况来对未来的肥胖程度进行评估,并根据评估结果寻求解决肥胖问题的合理方式。
2.2数据说明
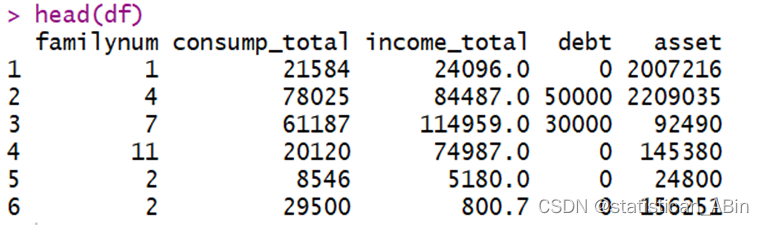
该数据集来自于UCL机器学习知识库,数据集包括墨西哥、秘鲁和哥伦比亚等国基于饮食习惯和身体状况的个体肥胖水平估计数据。数据包含17个属性和2111条记录,这些记录标有类变量肥胖等级,允许使用体重不足、正常体重、超重一级、超重二级、肥胖一级、肥胖二级和肥胖三级的值对数据进行分类。77%的数据是使用Weka工具和SMOTE过滤器综合生成的,23%的数据是通过网络平台直接从用户那里收集的。这些数据可用于生成智能计算工具,以识别个人的肥胖水平,并构建监控肥胖水平的推荐系统。部分原始数据如图。
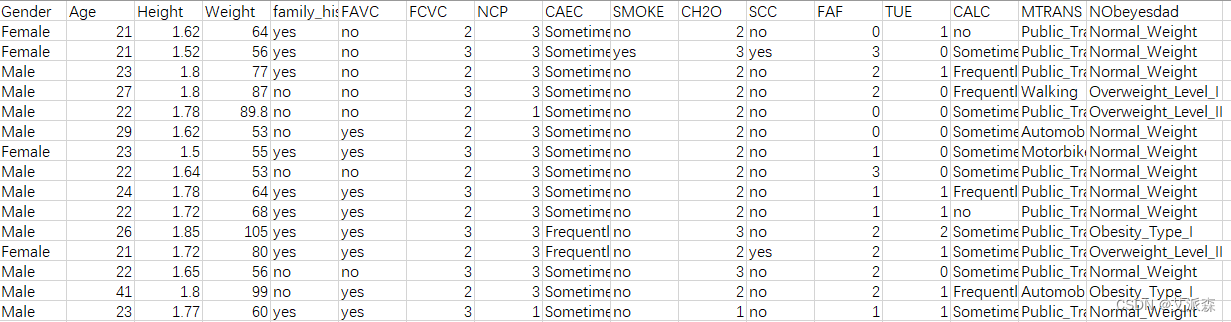
数据共包括17个属性,下面对各属性进行说明,如表 :
属性 |
含义 |
取值 |
Gender |
性别 |
Female、Male |
Age |
年龄 |
整数取值 |
Height |
身高 |
小数(m) |
Weight |
体重 |
整数取值(kg) |
Family history with overweight |
家庭肥胖历史 |
Yes、NO |
FAVC |
经常食用高热量食物 |
Yes、NO |
FCVC |
食用蔬菜的频率 |
No(0)、Sometimes(1)、Frequently(2)、Always(3) |
NCP |
主餐次数 |
1-2、3、>3 |
CAEC |
两餐之间食用食物 |
No、Sometimes、Frequently、Always |
SMOKE |
是否抽烟 |
Yes、NO |
CH2O |
每日饮水量 |
1(a little)、2(1-2L)、3(>2L) |
CALC |
饮酒 |
No、Sometimes、Frequently、Always |
SCC |
卡路里消耗监测 |
Yes、NO |
FAF |
身体活动频率 |
0(NO)、1(1-2天)、2(2-4天)、3(4-5天) |
TUE |
使用技术设备的时间 |
0(0-2h)、1(3-5h)、2(>5h) |
MTRANS |
使用的交通工具 |
Automobile、Motorbike、Bike、Public、Transportation、Walking |
NObeyesdad |
肥胖等级 |
Based on the WHO Classification |
Gender、Age、Family_history_with_overweight、FAVC、FCVC、NCP、CAEC、SMOKE、CH2O、CALC、SCC、FAF、TUE为特征, Height、Weight作为两个使用如下公式计算体重指数(MBI)的特征,NObeyesdad作为标签,计算出每个个体的体重指数后,为了确定肥胖水平,我们使用了世界卫生组织提供的表 ,对基于体重指数分析的数据进行了正确分类,得出每个个体的肥胖等级。
标准 |
等级 |
MBI <18.5 |
体重不足 |
18.5≤MBI≤24.9 |
正常 |
25.0≤MBI≤29.9 |
超重 |
30.0≤MBI≤34.9 |
肥胖一级 |
35.0≤MBI≤39.9 |
肥胖二级 |
MBI≥40 |
肥胖三级 |
2.3技术工具
Python版本:3.9
代码编辑器:jupyter notebook
3.算法原理
随机森林(Random Forest,RF),顾名思义就是将多棵相互之间并无关联的决策树整合起来形成一个森林,再通过各棵树投票或取均值来产生最终结果的分类器。在介绍随机森林前需要了解几个概念:Bootstrap 自助抽样法、Bagging 套袋法和 Boosting 提升法。
Bootstrap 是一种抽样方法,即采取随机有放回的方式采样数据,也就是每次抽取一个样本,再将其放回样本集中,下次还有可能抽到这个样本;而每轮中未抽到的数据组合起来,形成袋外数据集(Out of Band, OOB),用来在模型中做测试集。Bangging 和 Boosting 都是一种集成学习的方法,但两者有一些区别。Bagging 算法使用 Bootstrap 方法从原始样本集中随机不一定有放回的抽取n个样本,共抽取k轮,得到k个独立的训练集,元素可能有重复。每个训练集训练一个模型,得到k个结果,分类问题则从结果中取多数值作为最终结果,回归问题则取平均值作为最终结果。Boosting 则是对每个训练样本设立一个权值,被错分的样本在下一轮分类中会有更大的权值,也就是说,每轮样本相同但样本权重不同;对于分类器来说,分类误差小的拥有更大权值,分类误差大的相应权值更小。
随机森林采取的就是 bagging 方法,它将决策树用作 bagging 后的子分类模型。首先,对原始数据集使用 bootstrap 随机抽样的方法生成多个子训练集和相应的测试集,每个子训练集都构造一颗独立的决策树。其次,在构造决策树时,随机森林并不是在所有特征中找到性能最佳的特征进行分类,而是随机抽取一部分特征,在抽到的特征中间找到最优解应用于树节点进行分裂,这也是随机森林中两个关键随机步骤。最后由每个决策树投票产生最终的分类结果。随机森林由于有了 bagging,也就是集成的思想在,实际上相当于对样本和特征都进行了随机采样,所以可以避免过拟合。Bagging 策略过程如图(a)所示,随机森林过程如图(b)所示。
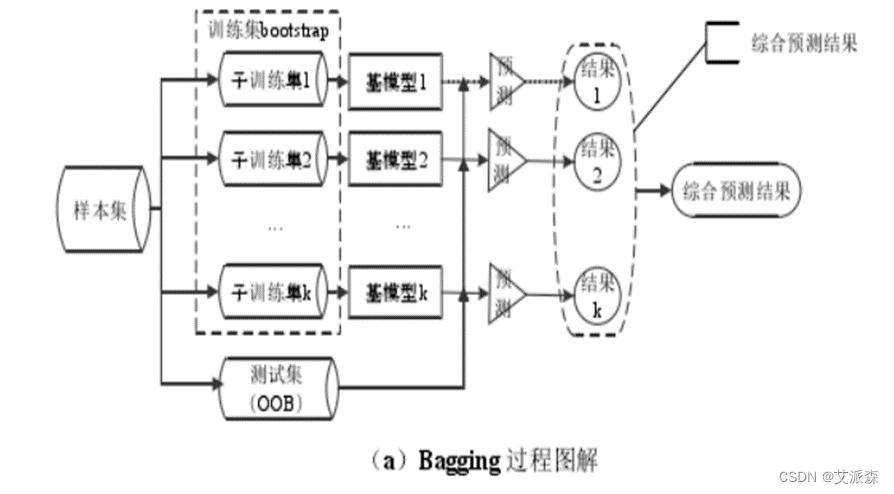
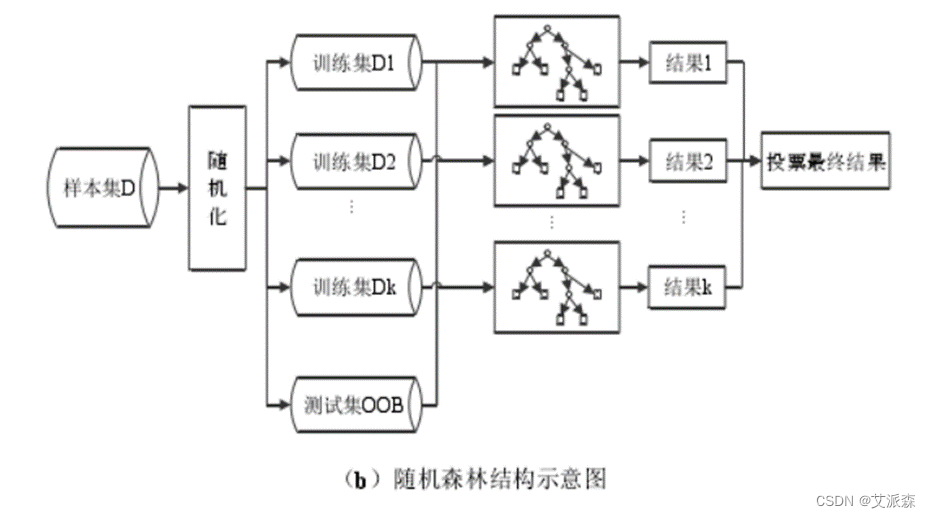
随机森林的构建过程用文字表述大致如下:假设原始样本集 D(X,Y),样本个数 n,要建立 k 棵树。
1) 抽取样本集:从原始训练集中随机有放回地抽取n个样本(子训练集)并重复n次,每一个样本被抽中的概率均为1/n。被剩下的样本组成袋外数据集(OOB),作为最终的测试集。
2) 抽取特征:从总数为 M 的特征集合中随意抽取m个组成特征子集,其中m<M。
3) 特征选择:计算节点数据集中每个特征对该数据集的基尼指数,选择基尼指数最小的特征及其对应的切分点作为最优特征与最优切分点(一般方法有 ID3、CART 和信息增益率),从节点生成两个子节点,将剩余训练数据分配到两个子节点中。
4) 生成 CART 决策树:在每个子节点的样本子集中重复执行3)步骤,递归地进行节点分割,直到生成所有叶节点。
5) 随机森林:重复执行(2)~(4),得到k棵不同的决策树。
6) 测试数据:每一棵决策树都对测试集中的每一条数据进行分类,统计 k 个分类结果,票数最多的类别,即为该样本的最终类别。
4.项目实施步骤
4.1理解数据
进行程序编写前先导入所需要的模块。

运行下面代码区域的代码以载入根据饮食习惯和身体状况估算肥胖水平的数据集,并输出数据类型,展示数据文件的前十行。

显示结果<class 'pandas.core.frame.DataFrame'>表示数据类型,输出的数据前十行如图所示:

4.2数据预处理
由于随机森林在学习数据规律进行预测时无法直接辨识字符对象,故需要对原始数据进行预处理,使用one_hot编码或者整数编码对原始的字符数据进行编码转化为机器学习可以识别的数字。
运行以下代码对原始数据进行整数编码,并显示数据前十行。

运行结果如图所示。

4.3探索性数据分析
下面将对各特征与标签之间的关系进行研究,通过下方代码绘制出年龄特征与标签的散点分布图关系,绘制结果如图 所示。


运行以下代码定义统计函数,从而对不同标签的同一特征进行统计。

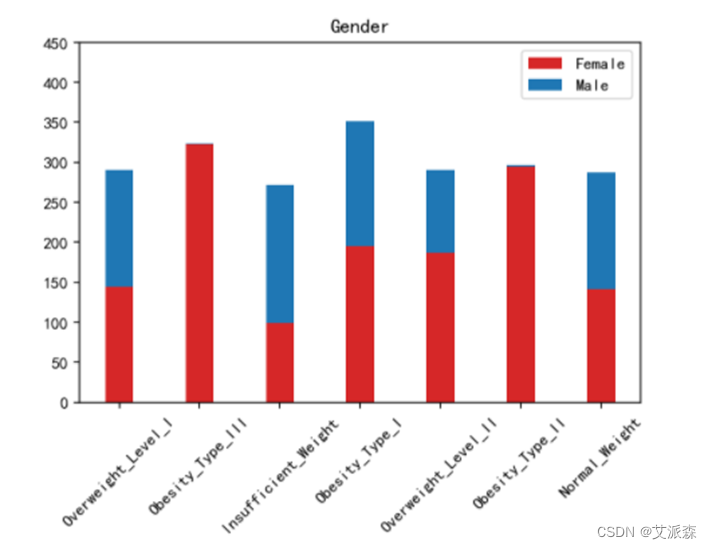
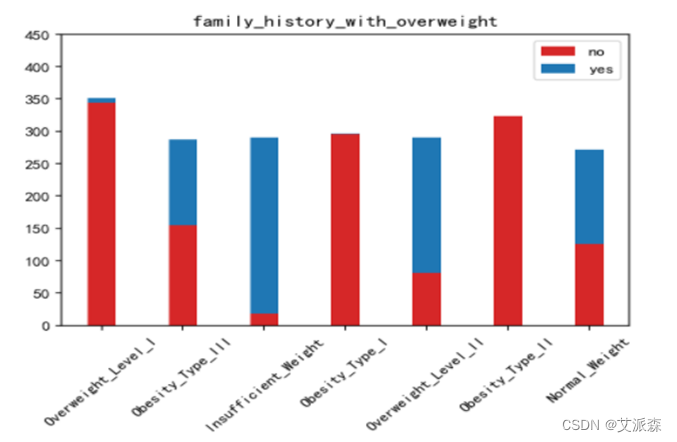
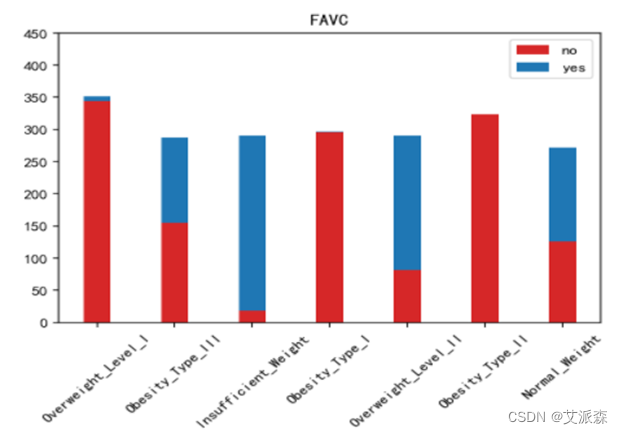
运行以下代码来绘制其他特征与标签之间的堆叠柱状图以探究他们之间是否存在潜在联系。(绘图代码原理相同不逐一罗列,只展示不同特征的绘图结果)。
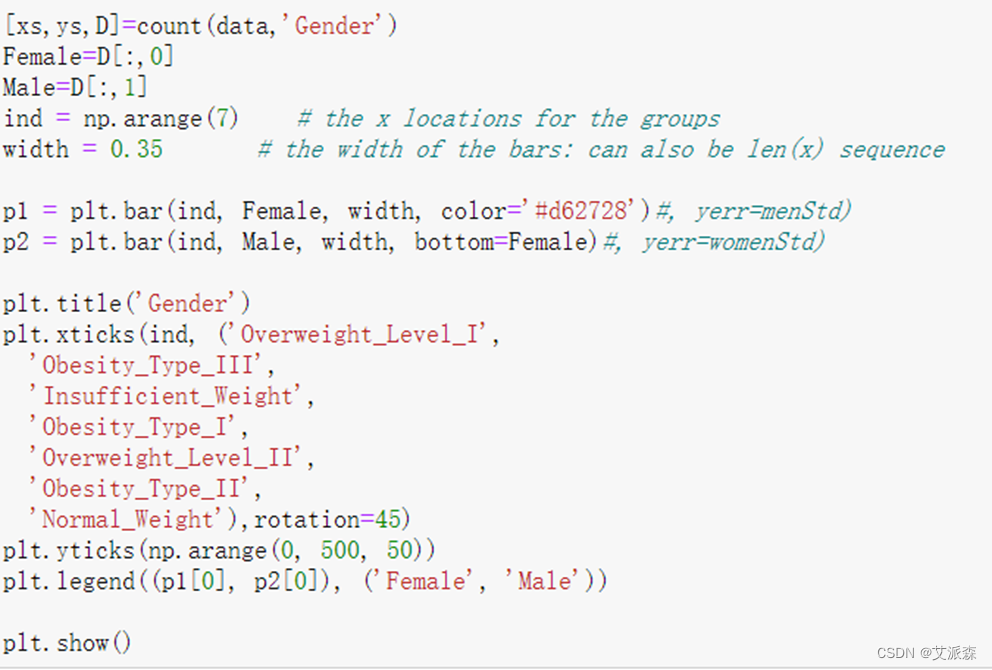
不同肥胖等级下的各特征的分布结果如下图所示:
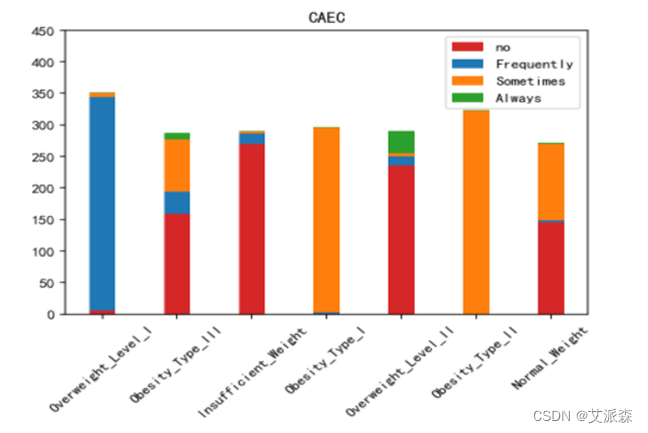
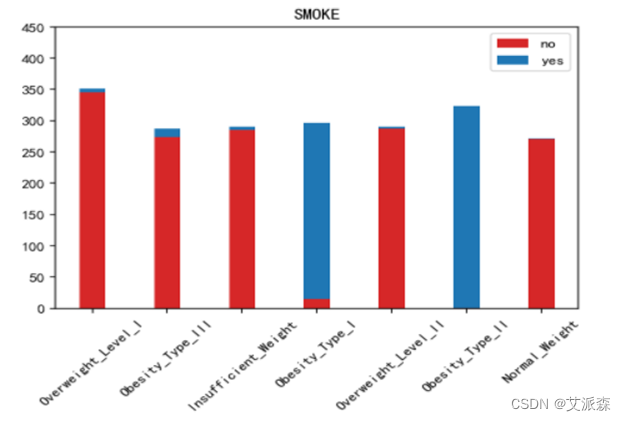
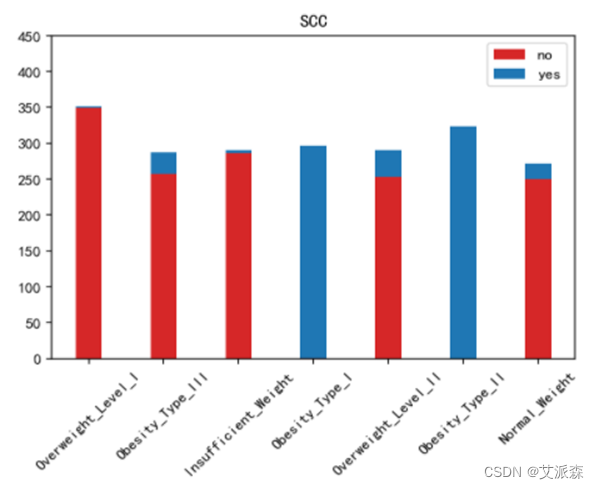
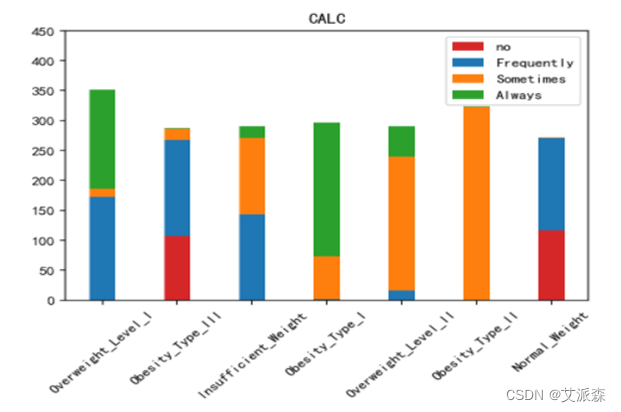

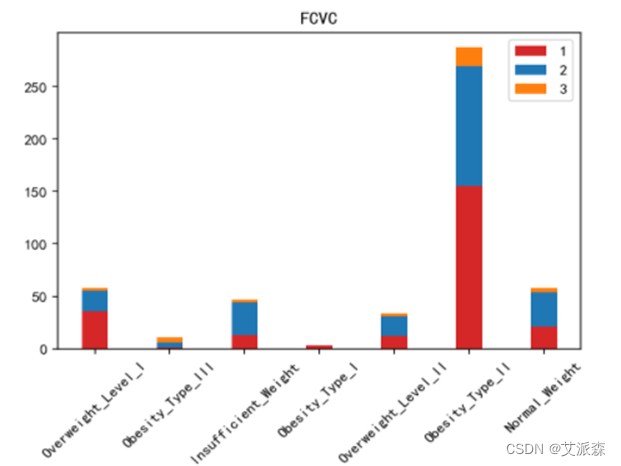
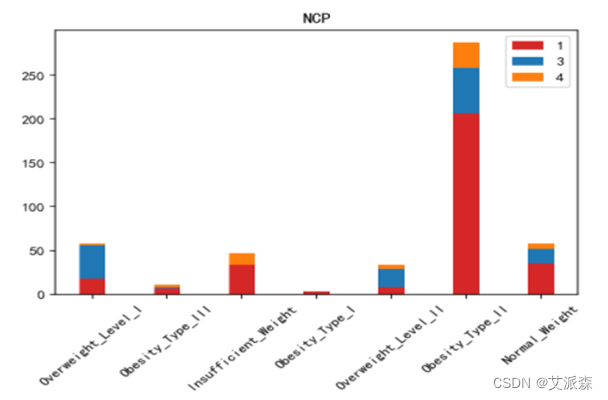
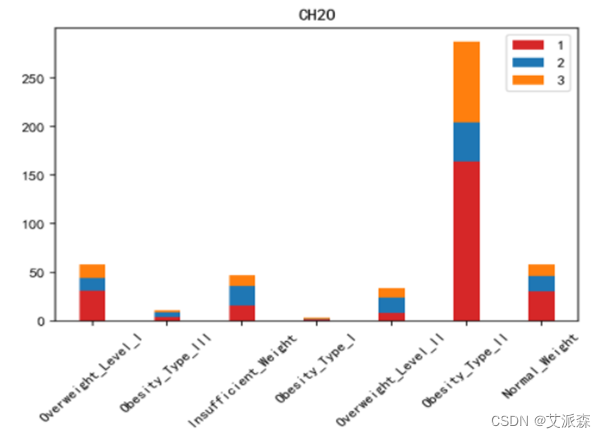

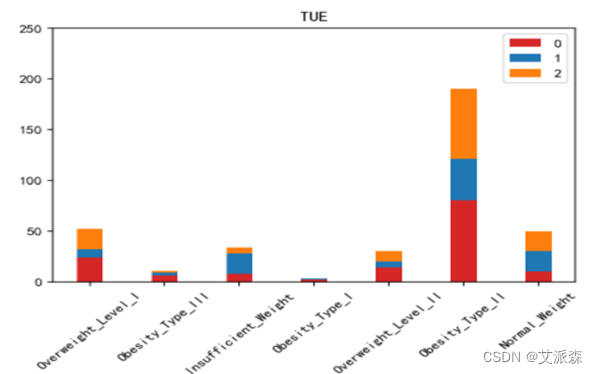
绘制热力图来查看特征与标签之间的相关性。


如上图所示,家族肥胖史与身体肥胖水平是强烈正相关,并与年龄以及是否经常食用高热量食物有较高的正相关性。两餐之间是否进食与身体肥胖水平是强烈负相关,并且与是否进行卡路里检测以及身体活动频率有较高负相关性。
4.4特征工程
模型参数分析
由于这个项目的最终目标建立一个预测肥胖成因的模型,我们需要将数据集分为特征子集(features)和标签子集(target variable)。
在如下代码中将原数据集的标签列提取至一个独立的列向量作为模型训练的表子集,并将剔除过标签列的数据集作为模型训练的特征子集。

在下面的代码中,需要
1)使用sklearn.model_ selection 中的train_ _test_ split,将features和prices的数据都分成用于训练的数据子集和用于测试的数据子集。其中70%的数据用于训练,30%用于测试;同时选定一个数值以设定train_ test_ split 中的random_ state,这会确保结果的一致性。
2)将分割后的训练集与测试集分配给X_train,X_test,y_train和 y_test。

运行结果为:

4.5模型构建
使用随机森林算法训练一个模型。为了得出的是一个最优模型,需要使用K折交叉验证训练模型,以找到最佳的n_estimators参数以及max_depth参数。n_estimators参数为森林中树的数目,n_estimators越大越好,但占用的内存与训练和预测的时间也会相应增长,且边际效益是递减的,所以要在可承受的内存/时间内选取尽可能大的n_estimators。而在sklearn中,n_estimators默认为10。max_depth参数为决策树最大深度。若等于None,表示决策树在构建最优模型的时候不会限制子树的深度。如果模型样本量多,特征也多的情况下,推荐限制最大深度;若样本量少或者特征少,则不限制最大深度。
运行以下代码,建立随机森林模型:
运行以上代码得到如图结果,运行结果可以得出模型的最大深度为10,决策树数量为100。

运行如下代码对模型进行训练。

训练完成后运行结果如图所示。

4.6模型评估
为了对模型进行评估,引用sklearn评估指标precision_score/recall_score/f1_score对模型的优良性进行评估。
评估模型的各项指标计算方法如下:
Relevant |
NonRelevant |
|
Retrived |
True Positive(TP) |
False Positive(FP) |
Not Retrived |
False Negative(FN) |
True Negative(TN) |
True/False:表示分类器分类是否正确。
Positive/Negative:表示分类器的分类结果。
TP:表示本来是正样例,分类成正样例(分类正确)。
FP:表示本来是负样例,分类成正样例(分类错误-错把负例分成正例)。
FN:表示本来是正样例,分类为负样例(分类错误-错把正例分成负例)。
TN:表示本来是负样例,分类成负样例(分类正确)。
P=TP+FP:表示被分类成正样例。
N=FN+TN:表示被分类成负样例。
1、准确率(accuracy):被分对的样本除以总样本数即
2、精确率(precision),也叫查准率:正样本识别正确的个数除以分类成正样本的个数即
,换句话说P度量的是识别为正样本的有多少本来是正样本。
3、召回率(recall),也叫查全率:正样本识别正确的个数除以本来是正样本的个数即
,换句话说R度量的是本来是正样本的有多少被识正确的识别为正样本。
4、F1值:因为P和R在某些情况下是矛盾的,比如在极端情况下,只搜出一个结果,且是准确的,则P=100%;但是R很低。需要综合考虑P和R。
运行以下代码对模型进行评估:
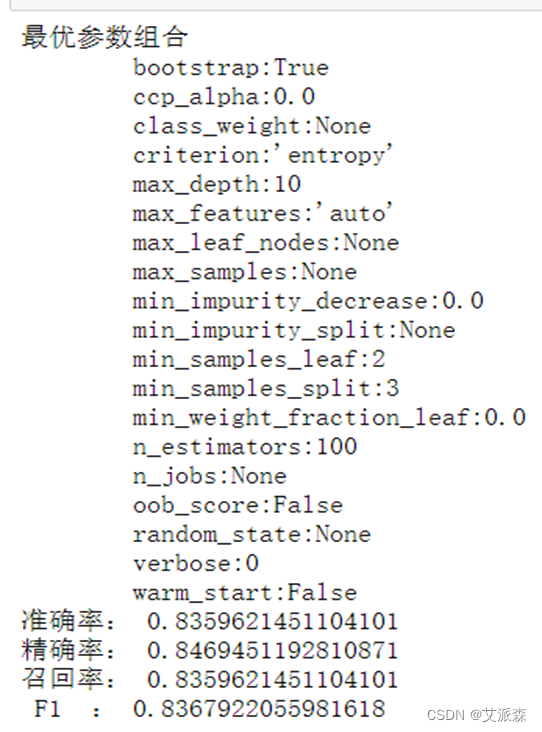
运行结果如图所示,可以的得到模型的准确率为83.6%,精确率为84.7%,召回率为83.6%,F1为83.7%。
为了更为直观的显示模型效果,运行下图代码绘制出分类报告表以及混淆矩阵图。

结果如图所示,从图中可以看出模型对于肥胖I型、肥胖II型、肥胖III型的预测准确率为100%,对于其余的体重指标预测有少量偏差,最低准确率为85%,但总体平均准确率有98%。
4.7模型预测
通过实际调查,获取三个人的饮食习惯和身体状况数据作为验证集如下表:
Number |
Gender |
Age |
Height |
Weight |
FHWO |
FAVC |
FCVC |
NCP |
1 |
Male |
32 |
1.65 |
80 |
Yes |
No |
2 |
2 |
2 |
Female |
25 |
1.55 |
49 |
No |
Yes |
3 |
3 |
3 |
Male |
42 |
1.74 |
94 |
Yes |
Yes |
3 |
2 |
Number |
CAEC |
SMOKE |
CH2O |
SCC |
FAF |
TUE |
CALC |
MTRANS |
1 |
Sometimes |
Yes |
1 |
Yes |
3 |
1 |
2 |
Public_Transportation |
2 |
No |
No |
3 |
Yes |
1 |
0 |
1 |
Walking |
3 |
Always |
Yes |
2 |
No |
3 |
2 |
3 |
Automobile |
根据表中被调查人员的饮食以及生活习惯利用本文的优化模型对其肥胖水平进行预测,运行下列代码对其进行预测。
 得出预测结果如图所示:
得出预测结果如图所示:

预测结果合理,有无家族肥胖史是肥胖成因中较为关键的因素,有家族肥胖历史的人较正常人更容易患肥胖症,年龄和饮食也对肥胖症有着重要的影响,年龄越大的人患有肥胖症的几率较高,两餐之间有加餐习惯同时长期使用高热量食品的人也有更高的几率患上肥胖症。但有规律的检测卡路里摄入检测以及较为频繁的身体活动也可以有效的降低肥胖症的发病几率。
验证集中的三个人,第一个人与第三个人都有肥胖家族病史,相对于第二个没有家族病史的人肥胖等级更高,可见有肥胖家族病史的人肥胖水平更高;第三个人相对于第一个人而言,年龄相对较大,也经常食用高热量食物,而且两餐之间有经常加餐的习惯,此外也没有进行卡路里消耗检测,相对于第一个人使用技术设备的时间也较长,饮酒频率也比较高,所以第三个人的肥胖水平更高,由此可见年龄、是否经常使用高热量食物、两餐之间加餐频率、是否进行卡路里消耗检测都对一个人的肥胖水平产生了重要的影响,而使用技术的时间、饮酒频率两个人之间差别不是特别大,但仍然对肥胖水平有一定的影响作用。
5.实验总结
肥胖是一种全球性的疾病,无论人们的社会或文化水平如何,它始终都是热点话题,而且全球患者的数量逐年增长。为了帮助对抗这种疾病,开发工具和解决方案去检测或预测疾病的出现显得非常重要,而数据挖掘是让我们发现信息的重要工具。
本文使用随机森林算法对数据集进行处理,通过对多个影响因子进行多标签分类获取各影响因子与肥胖水平之间的权值,由此建立肥胖评估模型,模型准确率达到83.6%,从而探究肥胖的成因。实验结果表明了众多影响因子与肥胖水平之间的关系,肥胖家族病史与肥胖水平之间强正相关,年龄以及是否经常食用高热量也与肥胖水平之间呈较强的正相关关系,也就是说,通常有肥胖家族病史的人患病可能性更大,年龄越大以及经常食用高热量食物的人更容易肥胖;是否进行卡路里消耗监测以及是否经常活动身体等与肥胖水平有着负相关关系,换言之,规律的监测卡路里消耗以及频繁的身体活动可以降低患病几率;是否频繁饮酒、长时间使用技术设备每日饮水量等对肥胖水平有一定影响。
因此,根据实验结果,要想控制肥胖应努力加强家庭可以采用的健康习惯,例如均衡白天的饮食、确定饮食时间、少吃高热量的食物、降低饮酒频率等;必须认识到,除了饮食变化外,增加日常体育活动,例如每天至少步行半小时,每天至少喝两升水,是必不可少的,因为没有不锻炼的饮食;对卡路里消耗进行规律检测,减少使用技术设备的时间等。儿童和成人的高肥胖率是导致总体肥胖率较高的原因,我们再也不能对此视而不见,应在生命早期阶段就进行预防和控制,这样才能可持续的解决肥胖问题,而我们每一个人也应该提高认识,养成健康的生活习惯。
随着云计算、物联网和移动互联网等技术的飞速发展,数据的类型和规模以前所未有的速度增长,而人工智能和数据挖掘的快速发展提高了数据管理效率。通过本实验对实际案例的研究与学习,对数据挖掘有关的知识有了初步的了解,为以后继续学习数据挖掘与分析奠定了基础。
因为对数据挖掘不够了解,实验过程中遇到了很多问题,感谢老师和同学的帮助。实验仍存在很多问题,如实验结果与实际情况存在偏差,模型准确率有待提高;算法的很多代码不够完善,存在漏洞;对实验结果分析不够深入,有待进一步挖掘等等。针对这些不足,在今后不断学习过程中会不断完善。
心得与体会:
通过这次Python项目实战,我学到了许多新的知识,这是一个让我把书本上的理论知识运用于实践中的好机会。原先,学的时候感叹学的资料太难懂,此刻想来,有些其实并不难,关键在于理解。
在这次实战中还锻炼了我其他方面的潜力,提高了我的综合素质。首先,它锻炼了我做项目的潜力,提高了独立思考问题、自我动手操作的潜力,在工作的过程中,复习了以前学习过的知识,并掌握了一些应用知识的技巧等
在此次实战中,我还学会了下面几点工作学习心态:
1)继续学习,不断提升理论涵养。在信息时代,学习是不断地汲取新信息,获得事业进步的动力。作为一名青年学子更就应把学习作为持续工作用心性的重要途径。走上工作岗位后,我会用心响应单位号召,结合工作实际,不断学习理论、业务知识和社会知识,用先进的理论武装头脑,用精良的业务知识提升潜力,以广博的社会知识拓展视野。
2)努力实践,自觉进行主角转化。只有将理论付诸于实践才能实现理论自身的价值,也只有将理论付诸于实践才能使理论得以检验。同样,一个人的价值也是透过实践活动来实现的,也只有透过实践才能锻炼人的品质,彰显人的意志。
3)提高工作用心性和主动性。实习,是开端也是结束。展此刻自我面前的是一片任自我驰骋的沃土,也分明感受到了沉甸甸的职责。在今后的工作和生活中,我将继续学习,深入实践,不断提升自我,努力创造业绩,继续创造更多的价值。
这次Python实战不仅仅使我学到了知识,丰富了经验。也帮忙我缩小了实践和理论的差距。在未来的工作中我会把学到的理论知识和实践经验不断的应用到实际工作中,为实现理想而努力。
源代码
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib as mpl
%matplotlib inline
import csv
import warnings
from numpy import newaxis
#使用高清显示
%config InlineBackend.figure_format = 'retina'
#加载文件
filename = 'ObesityDataSet_raw_and_data_sinthetic(1).csv'
data = pd.read_csv(filename,sep = ',',engine='python')
#data.head()
#数据的多行显示使用该函数, 也可以使用 raw.tail()从后向前显示
print(type(data))
print(data.head(10))#显示文件的前十行
#求各成分元素含量
def Content(values):
value_cnt = {} # 将结果用一个字典存储
# 统计结果
for value in values:
# get(value, num)函数的作用是获取字典中value对应的键值, num=0指示初始值大小。
value_cnt[value] = value_cnt.get(value, 0) + 1
# 打印输出结果
key = [key for key in value_cnt.keys()]
cnt = [value for value in value_cnt.values()]
cnt = np.array(cnt)
cnt1 = cnt/sum(cnt)
return key,cnt,cnt1
#年龄跟肥胖的数据分布
plt.rcParams['font.sans-serif']=['SimHei'] #显示中文标签
plt.rcParams['axes.unicode_minus']=False
Age = data["Age"]
NOb = data["NObeyesdad"]
plt.plot(Age,NOb,'o')
plt.xlabel('年龄')
plt.ylabel('肥胖状况')
plt.show()
data_gender = data[['Gender','Age','Height','Weight','family_history_with_overweight','FAVC','FCVC',
'NCP','CAEC','SMOKE','CH2O','SCC','FAF','TUE','CALC','MTRANS','NObeyesdad']]
data_gender_re = data_gender[data_gender.notnull()]
def count(data,X):
tj = data[X]
tq = data['NObeyesdad']
xs = list(set(tj))
ys = list(set(tq))
D =[]
for y in ys:
PP = data_gender_re.loc[(data_gender_re['NObeyesdad'] == y)]
PPN = PP[X]
cnt = []
for x in xs:
m = 0
for value in PPN:
if value == x :
m = m+1
cnt.append(m)
cnt = np.array(cnt)
D.append(cnt)
D=np.array(D)
return xs,ys,D
[xs,ys,D]=count(data,'Gender')
Female=D[:,0]
Male=D[:,1]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
plt.title('Gender')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0]), ('Female', 'Male'))
plt.show()
[xs,ys,D]=count(data,'family_history_with_overweight')
Female=D[:,0]
Male=D[:,1]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
plt.title('family_history_with_overweight')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0]), ('no', 'yes'))
plt.show()
[xs,ys,D]=count(data,'FAVC')
Female=D[:,0]
Male=D[:,1]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
plt.title('FAVC')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0]), ('no', 'yes'))
plt.show()
[xs,ys,D]=count(data,'CAEC')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
Male2=D[:,3]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
d1 = []
for i in range(0, 7):
sum = Female[i] + Male[i] + Female1[i]
d1.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
p4 = plt.bar(ind, Male2, width, bottom=d1)#, yerr=womenStd)
plt.title('CAEC')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0],p3[0],p4[0]), ('no', 'Frequently', 'Sometimes', 'Always'))
plt.show()
[xs,ys,D]=count(data,'SMOKE')
Female=D[:,0]
Male=D[:,1]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
plt.title('SMOKE')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0]), ('no', 'yes'))
plt.show()
[xs,ys,D]=count(data,'SCC')
Female=D[:,0]
Male=D[:,1]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
plt.title('SCC')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0]), ('no', 'yes'))
plt.show()
[xs,ys,D]=count(data,'CALC')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
Male2=D[:,3]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
d1 = []
for i in range(0, 7):
sum = Female[i] + Male[i] + Female1[i]
d1.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
p4 = plt.bar(ind, Male2, width, bottom=d1)#, yerr=womenStd)
plt.title('CALC')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0],p3[0],p4[0]), ('no', 'Frequently', 'Sometimes', 'Always'))
plt.show()
[xs,ys,D]=count(data,'MTRANS')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
Male2=D[:,3]
Male3=D[:,4]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
d1 = []
for i in range(0, 7):
sum = Female[i] + Male[i] + Female1[i]
d1.append(sum)
d2 = []
for i in range(0, 7):
sum = Female[i] + Male[i] + Female1[i] + Male2[i]
d2.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
p4 = plt.bar(ind, Male2, width, bottom=d1)#, yerr=womenStd)
p5 = plt.bar(ind, Male3, width, bottom=d2)#, yerr=womenStd)
plt.title('MTRANS')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 500, 50))
plt.legend((p1[0], p2[0],p3[0],p4[0],p4[0]), ('Automobile', 'Bike', 'Motorbike', 'Public_Transportation', 'Walking'))
plt.show()
#加载文件
filename1 = 'ObesityDataSet_raw_and_data_sinthetic.csv'
data1 = pd.read_csv(filename,sep = ',',engine='python')
[xs,ys,D]=count(data1,'FCVC')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
plt.title('FCVC')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 300, 50))
plt.legend((p1[0], p2[0],p3[0]), (1, 2, 3))
plt.show()
[xs,ys,D]=count(data1,'FAF')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
plt.title('FAF')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 300, 50))
plt.legend((p1[0], p2[0],p3[0]), (1, 3, 4))
plt.show()
[xs,ys,D]=count(data1,'CH2O')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
plt.title('CH2O')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 300, 50))
plt.legend((p1[0], p2[0],p3[0]), (1, 2, 3))
plt.show()
#[xs,ys,D]=count(data,'FAF')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
Male2=D[:,3]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
d1 = []
for i in range(0, 7):
sum = Female[i] + Male[i] + Female1[i]
d1.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
p4 = plt.bar(ind, Male2, width, bottom=d1)#, yerr=womenStd)
plt.title('FAF')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 300, 50))
plt.legend((p1[0], p2[0],p3[0],p4[0]), (0,1,2,3))
plt.show()
[xs,ys,D]=count(data1,'TUE')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
plt.title('TUE')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 300, 50))
plt.legend((p1[0], p2[0],p3[0]), (0, 1, 2))
plt.show()
[xs,ys,D]=count(data1,'NCP')
Female=D[:,0]
Male=D[:,1]
Female1=D[:,2]
ind = np.arange(7) # the x locations for the groups
width = 0.35 # the width of the bars: can also be len(x) sequence
d = []
for i in range(0, 7):
sum = Female[i] + Male[i]
d.append(sum)
p1 = plt.bar(ind, Female, width, color='#d62728')#, yerr=menStd)
p2 = plt.bar(ind, Male, width, bottom=Female)#, yerr=womenStd)
p3 = plt.bar(ind, Female1, width, bottom=d)#, yerr=womenStd)
plt.title('NCP')
plt.xticks(ind, ('Overweight_Level_I',
'Obesity_Type_III',
'Insufficient_Weight',
'Obesity_Type_I',
'Overweight_Level_II',
'Obesity_Type_II',
'Normal_Weight'),rotation=45)
plt.yticks(np.arange(0, 300, 50))
plt.legend((p1[0], p2[0],p3[0]), (1, 3, 4))
plt.show()
#文件预处理
#删除体重身高列
data=data.drop(["Height"],axis=1) #删除title这列数据
data=data.drop(["Weight"],axis=1)
gender1 = {'Male':1,'Female':2}
data["Gender"] = data["Gender"].map(gender1)
gender2 = {'yes':1,'no':0}
data["family_history_with_overweight"] = data["family_history_with_overweight"].map(gender2)
data["FAVC"] = data["FAVC"].map(gender2)
data["SMOKE"] = data["SMOKE"].map(gender2)
data["SCC"] = data["SCC"].map(gender2)
gender3 = {'no':0,'Sometimes':1,'Frequently':2,'Always':3}
data["CAEC"] = data["CAEC"].map(gender3)
data["CALC"] = data["CALC"].map(gender3)
gender4 = {'Bike':1,'Motorbike':2,'Public_Transportation':3,'Walking':4, 'Automobile':5}
data["MTRANS"] = data["MTRANS"].map(gender4)
gender5 = {'Insufficient_Weight':1, 'Normal_Weight':2, 'Obesity_Type_III':7, 'Overweight_Level_II':4, 'Obesity_Type_I':5, 'Overweight_Level_I':3, 'Obesity_Type_II':6}
data["NObeyesdad"] = data["NObeyesdad"].map(gender5)
print(data.head(10))
data.describe()#无实际意义
#相关性
"""
使用heatmap绘制相关性热力图
vmax设定热力图色块的最大区分值
square设定图片是否为正方形
annot 设定是否显示每个色块的系数值
"""
import seaborn as sns
fig = plt.figure(figsize=(18,18))
sns.heatmap(data.corr(),vmax=1)
plt.rcParams["font.sans-serif"] = ["Microsoft YaHei"] #解决plt乱码
plt.rcParams['axes.unicode_minus'] = False #解决plt乱码
from sklearn.model_selection import train_test_split
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import GridSearchCV
from sklearn.metrics import classification_report,confusion_matrix,\
precision_score, recall_score, accuracy_score,f1_score,roc_auc_score,\
roc_curve,auc,explained_variance_score
#构造模型数据集
data3 = data.drop(['NObeyesdad'],axis = 1)
y_1 = data['NObeyesdad']
#进行训练集与测试集的划分
data3.shape
X = data3.iloc[:,0:75].values
y = y_1.values
X_train,X_test,y_train,y_test = train_test_split(X,y,test_size= 0.3,random_state=1)
#建立随机森林模型
from sklearn.ensemble import RandomForestRegressor
from sklearn.metrics import confusion_matrix,log_loss
import time
import numpy as np
range_n = np.logspace(0,2,num=5).astype(int)
range_n #树的深度
#通过树的大小和K折验证得到log_loss最小的值和最优树的数量
# find the best n_estimators for RandomForestClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import KFold
print('Finding best n_estimators for RandomForestClassifier...')
min_score = 100000
best_n = 0
scores_n = []
for n in range_n:
print("the number of trees : {0}".format(n))
t1 = time.time()
rfc_score = 0.
rfc = RandomForestClassifier(n_estimators=n)
kf = KFold(n_splits=3, shuffle=False, random_state=1)
for train_k, test_k in kf:
rfc.fit(X_train.iloc[train_k], y_train.iloc[train_k])
#rfc_score += rfc.score(train.iloc[test_k], train_y.iloc[test_k])/10
pred = rfc.predict(X_train.iloc[test_k])
rfc_score += log_loss(y_train.iloc[test_k], pred) / 10
scores_n.append(rfc_score)
if rfc_score < min_score:
min_score = rfc_score
best_n = n
t2 = time.time()
print('Done processing {0} trees ({1:.3f}sec)'.format(n, t2-t1))
print(best_n, min_score)
# find best max_depth for RandomForestClassifier
print('Finding best max_depth for RandomForestClassifier...')
min_score = 100000
best_m = 0
scores_m = []
range_m = np.logspace(0,2,num=3).astype(int)
for m in range_m:
print("the max depth : {0}".format(m))
t1 = time.time()
rfc_score = 0.
rfc = RandomForestClassifier(max_depth=m, n_estimators=best_n)
for train_k, test_k in KFold(len(train_kobe), n_splits=10, shuffle=True):
rfc.fit(X_train.iloc[train_k], y_train.iloc[train_k])
#rfc_score += rfc.score(train.iloc[test_k], train_y.iloc[test_k])/10
pred = rfc.predict(X_train.iloc[test_k])
rfc_score += log_loss(y_train.iloc[test_k], pred) / 10
scores_m.append(rfc_score)
if rfc_score < min_score:
min_score = rfc_score
best_m = m
t2 = time.time()
print('Done processing {0} trees ({1:.3f}sec)'.format(m, t2-t1))
print(best_m, min_score)
#使用决策树算法
from sklearn import tree
clf = GridSearchCV(estimator=tree.DecisionTreeClassifier(),
param_grid=[{'criterion':('gini','entropy'),
'splitter':('best','random'),
'max_depth':range(3,8,1),
'min_samples_split':range(2,11,1),
'min_samples_leaf':range(2,11,1)}],
n_jobs = -1,
cv = 5)
#使用随机森林
from sklearn.ensemble import RandomForestClassifier
clf = GridSearchCV(estimator=RandomForestClassifier(),
param_grid=[{'n_estimators':range(10,100,10),
'criterion':('gini','entropy'),
'max_depth':range(3,8,1),
'min_samples_split':range(2,11,1),
'min_samples_leaf':range(2,11,1)}],
n_jobs = -1,
cv = 5)
clf.fit(X_train,y_train)
# 预测结果
y_pred_array = clf.predict(X_test)
y_pred_prob =clf.predict_proba(X_test)
print('最优参数组合')
best_parameters=clf.best_estimator_.get_params()
for param_name in sorted(best_parameters.keys()):
print('\t%s:%r' %(param_name,best_parameters[param_name]))
print('准确率:',accuracy_score(y_test,y_pred_array))
print('精确率:',precision_score(y_test,y_pred_array,average='weighted'))
print('召回率:',recall_score(y_test,y_pred_array,average='weighted'))
print(' F1 :',f1_score(y_test,y_pred_array,average='weighted'))
#模型效果
print(classification_report( y_true=y_test, y_pred=y_pred_array ))
#画出混淆矩阵图
confmat = confusion_matrix( y_true=y_test, y_pred=y_pred_array )
fig,ax = plt.subplots(figsize=(2.5,2.5))
ax.matshow(confmat,cmap=plt.cm.Blues,alpha=0.3)
for i in range(confmat.shape[0]):
for j in range(confmat.shape[1]):
ax.text(x=j,y=i,s=confmat[i,j],va='center',ha='center')
plt.xlabel('预测值')
plt.ylabel('实际值')
plt.show()
#生成三组数据
fat_data=[[1,32,1,0,3,2,1,1,2,1,3,1,3,3],
[2,25,0,1,2,3,0,0,1,1,1,0,1,4],
[1,42,1,1,1,2,3,1,3,0,2,2,2,5]]
#进行预测
fat = clf.predict(fat_data)
for i,fat in enumerate(fat):
print("The weight index of interviewee" +str(i)+ "is fat"+str(fat))