介绍PLUMED
PLUMED 是一个插件,可以处理大量分子动力学代码。它可用于即时分析动力学特征或执行各种自由能方法。PLUMED 还可以用作,对以大多数现有格式保存的轨迹执行分析。
接口代码
- Amber, pmemd module, since version 20.
- AmberTools, sander module, since version 15.
- CP2K, since Feb 2015.
- ESPResSo, in a version that has been patched with PLUMED can be found here.
- PINY-MD, in its plumed branch.
- IPHIGENIE.
- AceMD, see this link.
- OpenMM, using the openmm-plumed plugin.
- DL_POLY4.
- VNL-ATK, see this link.
- ABIN.
- i-pi.
- LAMMPS since Nov 2018.
- Yaff, since Jul 2019.
- DFTB+, since release 20.1.
- Metalwalls
- ASE
- GPUMD
- gromacs-2020-7
- gromacs-2021-7
- gromacs-2022-5
- gromacs-2023
- namd-2-12
- namd-2-13
- namd-2-14
- qespresso-5-0-2
- qespresso-6-2
- qespresso-7-0
- qespresso-7-2
进行第一个元动力学训练
首先注释这个plumed命令
plumed.dat
MOLINFO STRUCTURE=diala.pdb
激活MOLINFO功能:系统中分子的信息可以以 pdb 文件的形式提供,也可以作为一组描述系统中各种链的原子列表提供。如果使用 pdb 文件
如果您使用的是 gromacs,最安全的方法是使用
gmx editconf -f topol.tpr -o reference.pdbphi: TORSION ATOMS=__FILL__
psi: TORSION ATOMS=__FILL__
计算扭转角。此命令可用于计算四个原子之间的扭转,或者用于计算投影在与轴正交的平面上的两个矢量之间的角度。
例子:
metad: METAD ARG=phi PACE=500 HEIGHT=1.2 BIASFACTOR=1 SIGMA=0.2,0.2 FILE=HILLS
在phi中激活良好调节的元动力学;每500个时间步沉积一个高斯函数,初始高度为1.2 kJ/mol;应该明智地选择偏差因子;
#高斯宽度(sigma)应根据无偏运行中的CV波动来选择;#高斯将被写入文件,也存储在网格中
PRINT ARG=phi,psi FILE=COLVAR STRIDE=10
每10步在COLVAR文件上打印两个集合变量
运行命令
gmx mdrun -s topol.tpr -nsteps 5000000 -plumed plumed.dat
运行完成后分析结果:
运行完成后,会产生一下几个文件:
- COLVAR:包含
PRINT命令指定的所有信息 - HILLS:包含沿模拟放置的高斯图的列表
- confout.gro
- ener.edr
- md.log
- state.cpt
- traj_comp.xtc
在元动力学模拟过程中,PLUMED将创建两个名为COLVAR和HILLS的文件。COLVAR文件包含由PRINT命令指定的所有信息,在这种情况下,骨干dihedrals φ和ψ的值每10步模拟。我们可以使用gnuplot来可视化模拟过程中元动力学CV φ的行为:
HILLS文件:模拟时间、phi和psi的值、phi和psi的高斯宽度、高斯高度和偏移因子
偏移因子的大小仅与回火元动力学模拟相关(请参见 Exercise 4. Setup and run a well-tempered metadynamics simulation, part I),它在标准元动力学模拟中等于1。我们稍后将使用HILLS文件来计算元动力学模拟的自由能,并评估其收敛性。目前,我们可以绘制模拟过程中CV的变化:


(没观察到)通过上图我们可以看到,系统初始为丙氨酸二肽的两种亚稳态之一。一段时间后(t=0.3ns),系统被元动力学偏置势推动,访问了另一个局部最小值。随着模拟的继续,偏置势填充了底层的自由能景观,系统能够在整个相空间中扩散。
Standard metadynamics simulation with grid
如果我们使用上述 PLUMED 输入文件,元动力学模拟的费用会随着模拟的长度而增加,因为在每一步都必须评估越来越多的高斯值。为了避免这个问题,您可以将偏差存储在网格上。为了使用网格,我们必须在METAD指令的行中添加一些附加信息,
# 定义 Phi 和 Psi 二面角设置
phi: TORSION ATOMS=5,7,9,15
psi: TORSION ATOMS=7,9,15,17
#
# 激活使用 phi 和 psi 的元动力学
# 每 500 个时间步长进行一次高斯沉积,
# 高度等于 1.2 kJ/mol,
# 两个CV的宽度(标准差)均为 0.35 rad
# 偏置势将会储存到网格中
# 对于两个CV而言,网格窗口大小均为 0.1 rad
# 对于两个CV而言,网格边界均为 -pi and pi
#
METAD ...
LABEL=metad
ARG=phi,psi
PACE=500
HEIGHT=1.2
SIGMA=0.35,0.35
FILE=HILLS
GRID_MIN=-pi,-pi
GRID_MAX=pi,pi
GRID_SPACING=0.1,0.1
... METAD
# 监测这两个变量和元动力学的偏置势
PRINT STRIDE=10 ARG=phi,psi,metad.bias FILE=COLVAR
Exercise 2. Restart a metadynamics simulation
如果我们尝试在已经存在COLVAR和HILLS文件的目录中使用上面的脚本再次运行元动力学模拟,PLUMED将创建旧文件的备份副本,并运行新的模拟。相反,如果我们想重新启动以前的模拟,我们必须将关键字RESTART添加到 PLUMED 输入文件中,如下所示:
# restart previous simulation
RESTART# set up two variables for Phi and Psi dihedral angles
phi: TORSION ATOMS=5,7,9,15
psi: TORSION ATOMS=7,9,15,17
#
# Activate metadynamics in phi and psi
# depositing a Gaussian every 500 time steps,
# with height equal to 1.2 kJoule/mol,
# and width 0.35 rad for both CVs.
#
metad: METAD ARG=phi,psi PACE=500 HEIGHT=1.2 SIGMA=0.35,0.35 FILE=HILLS# monitor the two variables and the metadynamics bias potential
PRINT STRIDE=10 ARG=phi,psi,metad.bias FILE=COLVAR
同时 Gromacs 也进行续跑:
gmx convert-tpr -s topol.tpr -extend 1000 -o topol1.tpr
gmx mdrun -v -deffnm topol1 -cpi state.cpt -plumed plumed.dat -noappend
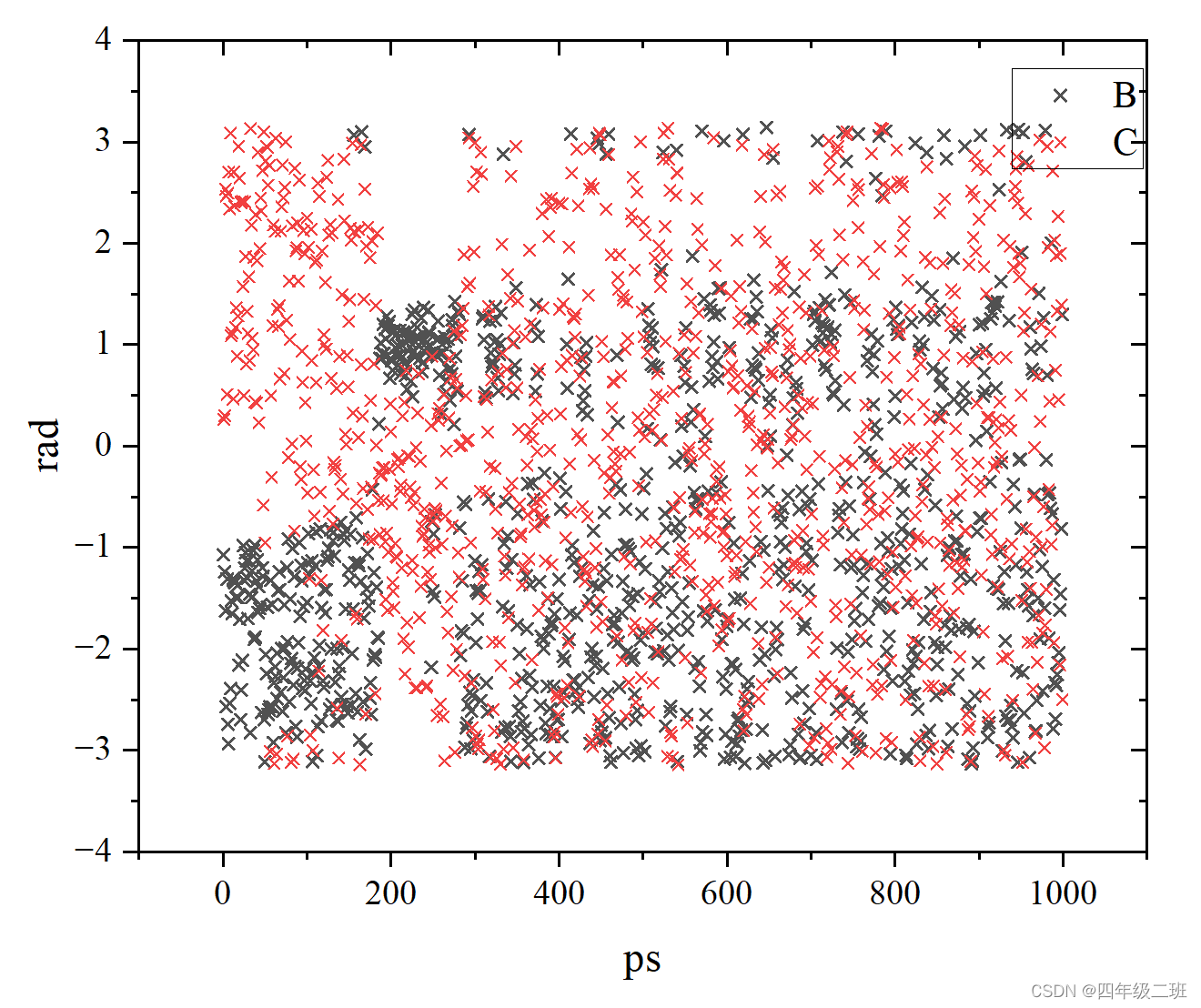
延长时间可以观察到上述的结果特征
通过这种方式,PLUMED 将从HILLS文件中读取旧的高斯值,并将新信息附加到COLVAR和HILLS两个文件中。
Exercise 3. Calculate free-energies and monitor convergence
Two-dimensional free energy
可以直接根据元动力学偏置势来估计自由能作为元动力学CV的函数。为了做到这一点,应使用sum_hills程序对模拟过程中存储并存储在HILLS文件中的高斯进行求和。为了计算作为phi和psi函数的二维自由能,使用以下命令行就足够了:
plumed sum_hills --hills HILLS
上面的命令生成一个名为fes.dat的文件,内含规则网格上计算的作为phi和psi函数的自由能表面。通过使用sum_hills的以下选项,可以修改自由能量文件的默认名称,以及网格的边界和窗口大小:
--outfile # 设置输出文件路径/名称
--min # 网格的最小边界
--max # 网格的最大边界
--bin # 网格的窗口数量
--spacing #网格间距,与窗口数量等效
😇对于输出文件fes.dat,译者发现qtgrace等画图软件似乎没法很好的绘制三维形貌图。所以这里给一个MATLAB绘制的方法:1. 将fes.dat中的前三列导入MATLAB中;2. 格栅化[X,Y,Z]=griddata(CV1,CV2,FES,linspace(min(CV1),max(CV1))',linspace(min(CV2),max(CV2)));3. 绘制三维图mesh(X,Y,Z) 或等高线图contour(X,Y,Z,20)😇
三维图mesh(X,Y,Z)

等高线图contour(X,Y,Z,20)

One-dimensional free energy
还可以从二维元动力学模拟中计算一维自由能。例如,如果对自由能单独作为 phi 二面角的函数感兴趣,则应使用以下命令行:
plumed sum_hills --hills HILLS --idw phi --kt 2.5
通过matlab画图
>> plot(a(:,1),a(:,2))
>> xlabel('phi [rad]')
>> ylabel('FES [kJmol/mol]')

Monitor convergence
为了评估元动力学模拟的收敛性,可以计算自由能的估计值作为模拟时间的函数。在收敛时,除了恒定的偏移之外,重构的轮廓应该是相似的。选项–stride应用于估计每N个高斯沉积的自由能,选项–mintozero可用于通过将全局最小值设置为零来对齐轮廓。使用以下命令行,可得下图左:
plumed sum_hills --hills HILLS --idw phi --kt 2.5 --stride 500 --mintozero
产生三个文件:fes_0.dat, fes_1.dat, fes_2.dat
用MATLAB画图
>> plot(b(:,1),b(:,2),'r')
>> hold on
>> plot(b(:,3),b(:,4),'g')
>> plot(b(:,5),b(:,6),'b')
>> xlabel('phi [rad]')
>> ylabel('FES [kJmol/mol]')

为了更定量地评估模拟的收敛性,我们可以计算一维自由能中沿 phi 的两个局部极小值之间的自由能差,作为模拟时间的函数。我们可以使用bash脚本analyze_FES.sh来积分两个盆地中的多个自由能剖面,这两个盆地由 phi 空间中的以下区间定义:basin A, -3<phi<-1, basin B, 0.5<phi<1.5;运行下行命令,可得上图右:
./analyze_FES.sh NFES -3.0 -1.0 0.5 1.5 KBT实际: ./analyze_FES.sh 3 -2.0 -1.0 0.5 1.5 2.5

其中,NFES是由sum_hills的步长选项生成的轮廓数(模拟不同时间的自由能量估计),KBT是以能量单位表示的温度(在这种情况下,KBT=2.5)。
以上所有分析及 Ex1. 对CV空间中扩散行为的观察表明,模拟是收敛的。
Exercise 4. Setup and run a well-tempered metadynamics simulation, Part I
在本练习中,我们将使用两个主链二面角 phi 和 psi 作为CV,在真空中对丙氨酸二肽进行良好的元动力学模拟。要使用回火元动力学,我们需要在METAD的行中添加两个关键字,该行指定模拟的偏置因子和温度。对于第一个例子,我们将尝试一个等于6的偏置因子。以下是 PLUMED 输入文件的内容:
plumed.dat
MOLINFO STRUCTURE=diala.pdb
# set up two variables for Phi and Psi dihedral angles
phi: TORSION ATOMS=5,7,9,15
psi: TORSION ATOMS=7,9,15,17
#
# Activate metadynamics in phi and psi
# depositing a Gaussian every 500 time steps,
# with height equal to 1.2 kJoule/mol,
# and width 0.35 rad for both CVs.
#
metad: METAD ARG=phi,psi PACE=500 HEIGHT=1.2 SIGMA=0.35,0.35 FILE=HILLS BIASFACTOR=6.0 TEMP=300.0# monitor the two variables and the metadynamics bias potential
PRINT STRIDE=10 ARG=phi,psi,metad.bias FILE=COLVAR
运行命令:
gmx mdrun -s topol.tpr -nsteps 500000 -plumed plumed.dat
在使用上述指令运行模拟之后,我们可以查看HILLS文件(如下)。与标准元动力学不同,HILLS文件的最后两列报告了更多有用的信息。最后一列包含所使用的偏置因子的值,而倒数第二列包含高斯高度,高斯在模拟过程中会按照回火方法重新缩放。

如果我们仔细查看高度列,我们会注意到在开始时报告的值高于输入文件中指定的初始高度,该初始高度应为1.2 kJ/mol。事实上,这一列报道了由预因子重新缩放的高斯高度,该预因子在回火元动力学中将偏置势与自由能联系起来。这样,当我们使用sum_hills时,沉积的高斯数之和将直接提供自由能,而无需进一步重新缩放。
我们可以绘制CV和高斯高度随时间的演变(偏置因子为 6):
在HILLS文件第一二三行,CV随着时间的演变

在HILLS文件第一六行,高斯高度随时间的演变(计算时间太短了)

按照前面例子中为标准元动力学描述的程序,我们可以估计自由能作为时间的函数,并使用analyze_FES.sh脚本监测模拟的收敛性。我们将对偏置因子设置为 6.0 的模拟执行此操作。在这种情况下,我们会注意到(如下图),在标准元动力学中观察到的振荡在这里是阻尼的,并且偏置势更平滑地收敛到潜在的自由能景观,前提是偏置因子足够高,可以跨越所研究系统的自由能垒。
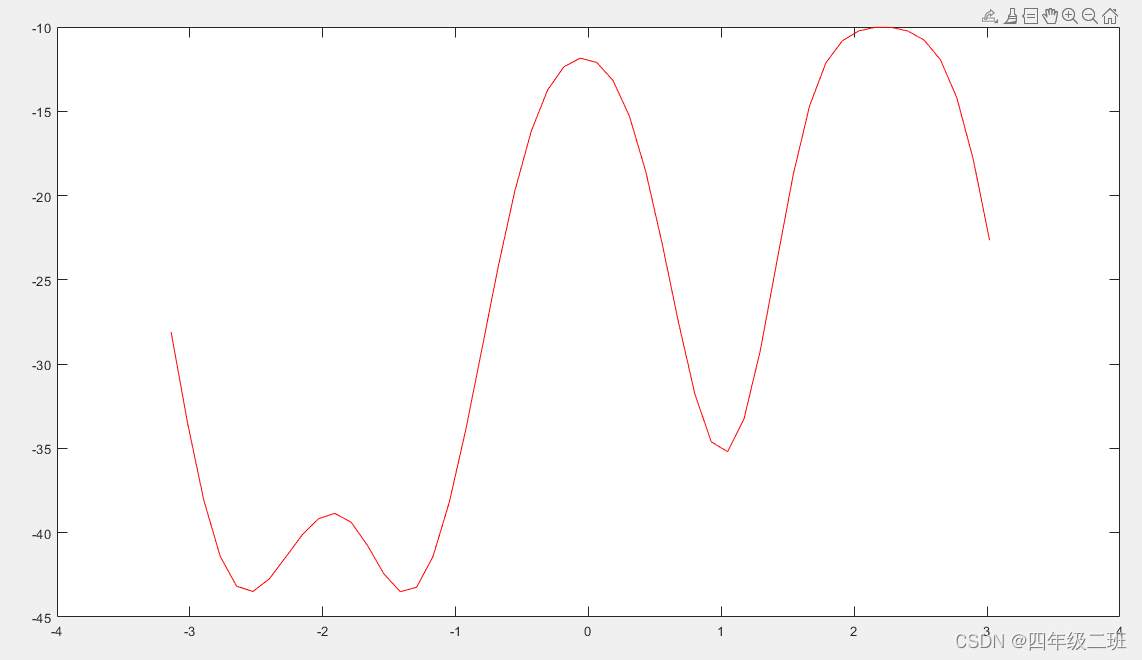
按照前面例子中为标准元动力学描述的程序,我们可以估计自由能作为时间的函数,并使用analyze_FES.sh脚本监测模拟的收敛性。我们将对偏置因子设置为 6.0 的模拟执行此操作。在这种情况下,我们会注意到(如下图),在标准元动力学中观察到的振荡在这里是阻尼的,并且偏置势更平滑地收敛到潜在的自由能景观,前提是偏置因子足够高,可以跨越所研究系统的自由能垒。
(由于机器受限,所以计算时间有点短)

Exercise 5. Setup and run a well-tempered metadynamics simulation, Part II
在本练习中,我们将研究在选择元动力学 CV 时忽略相关自由度的影响。我们将使用以下 PLUMED 输入文件,以 psi 二面体单独作为 CV 来运行一个回火元动力学模拟:
# set up two variables for Phi and Psi dihedral angles
phi: TORSION ATOMS=5,7,9,15
psi: TORSION ATOMS=7,9,15,17
#
# Activate metadynamics in psi
# depositing a Gaussian every 500 time steps,
# with height equal to 1.2 kJoule/mol,
# and width 0.35 rad.
# Well-tempered metadynamics is activated,
# and the biasfactor is set to 10.0
#
metad: METAD ARG=psi PACE=500 HEIGHT=1.2 SIGMA=0.35 FILE=HILLS BIASFACTOR=10.0 TEMP=300.0# monitor the two variables and the metadynamics bias potential
PRINT STRIDE=10 ARG=phi,psi,metad.bias FILE=COLVAR
运行命令(5ns)
gmx mdrun -s topol.tpr -nsteps 2500000 -plumed plumed.dat
查看COLVAR

从上面的图中可以清楚地看出,在 t=3ns 左右发生的事情是被忽视的慢变量 phi 从一个自由能池跳到另一个自由能量池。phi 的动力学不受任何势的影响,因此我们需要平衡这一自由度,即在模拟收敛之前,观察两个盆地的多次转换。或者,我们可以将 phi 添加到 CV 集。该示例演示了如何确认回火的元动力学模拟的收敛性——有必要但不足以观察到:1)高度非常小的高斯,2)CV空间中的扩散行为(如本示例的前3ns)。
我们应该做的是从不同的初始构象开始多次重复模拟。如果在所有模拟中,当高斯高度很小时,我们在偏置 CV 中观察到扩散行为,并且我们获得了非常相似的自由能表面,那么我们可以非常确信我们的模拟收敛到了正确的值。如果不是这样,手动检查运行结果(轨迹)可以帮助我们确定缺少的慢速自由度,以便添加到有偏差的CV集中。
结束!!!

































![[AIGC] Spring Boot中的切面编程和实例演示](https://img-blog.csdnimg.cn/direct/81f7c118fc3446be9436689c1c2832dd.png)

