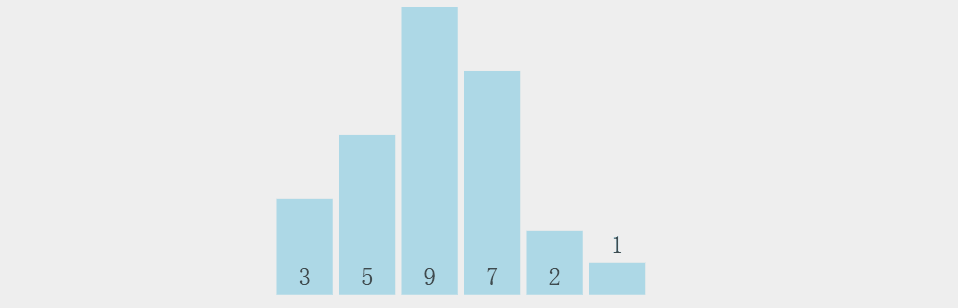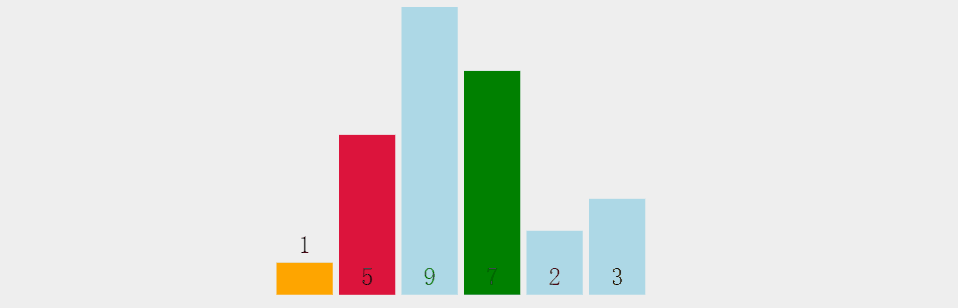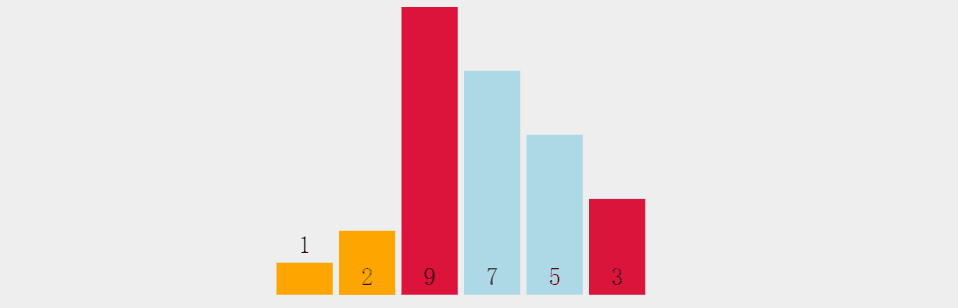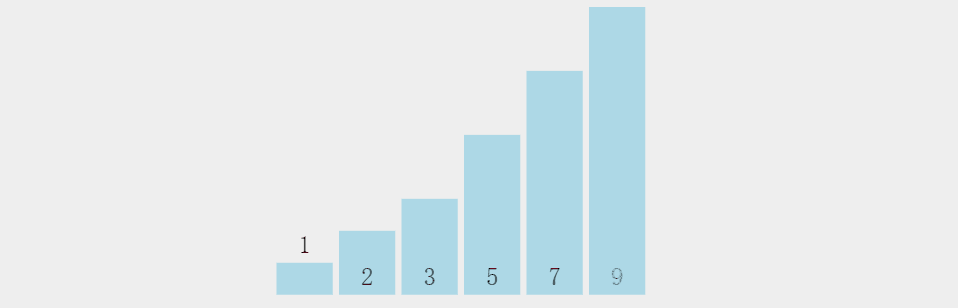文章目录
一、基于DeepSlice的切片配准
- 中文翻译:DeepSlice: 快速全自动配准小鼠脑成像到体积图谱
- 论文地址:DeepSlice: rapid fully automatic registration of mouse brain imaging to a volumetric atlas
- github源码:https://github.com/PolarBean/DeepSlice
- web界面:https://www.deepslice.com.au/

1.1、研究现状
当前现状
- 精准配准的难易程度:依赖于专业知识,需要具备神经解剖学知识;
- 现有工具的使用条件:大数据的图像处理、高水平的编程能力、高耗时的脑区处理。
- 卷积神经网络CNN:在影像数据的自动分析(包括细胞组织学和姿态估计)中显示出了巨大的前景,但目前还没有用于将切片组织配准到体积图谱的等效工具。
基于上述问题,论文提出了将冠状小鼠的脑切片自动向CCF对齐的深度神经网络(DeepSlice)。
1.2、网络模型(DeepSlice)
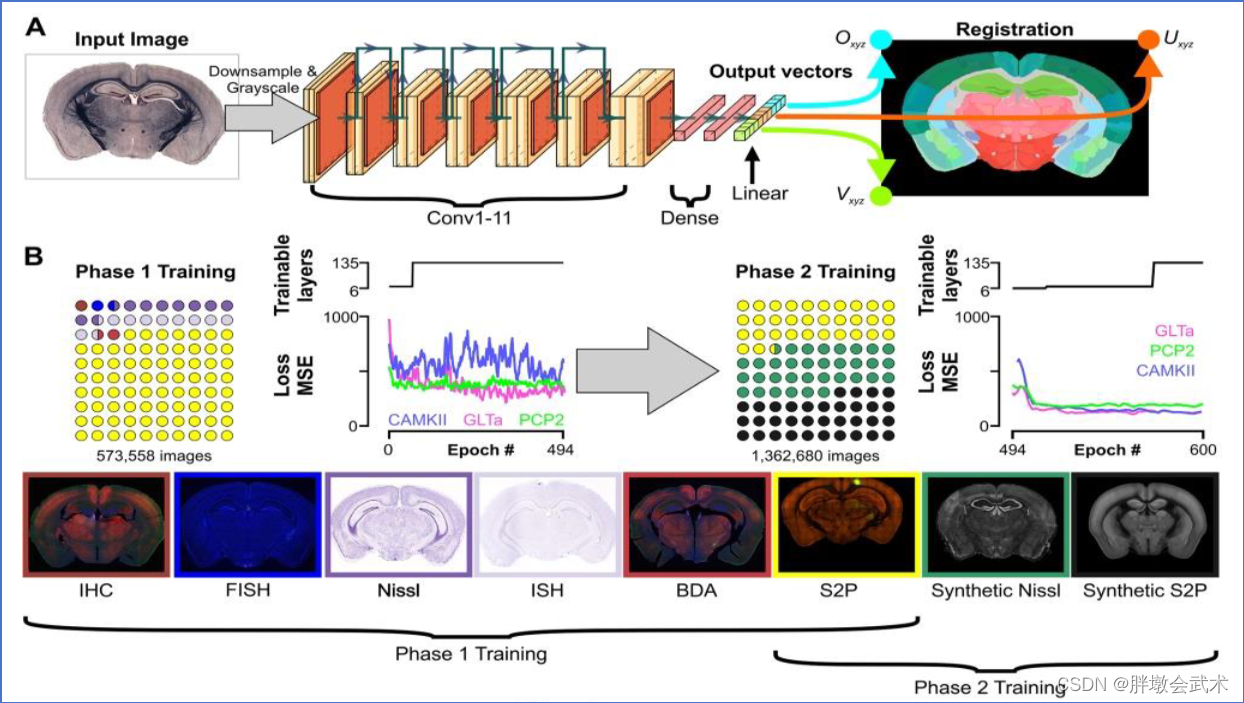
- DeepSlice是一个构建在 Xception 基础上的CNN模型,通过移除 Xception 最后一层的Softmax,并替换为2个Dense层 + 1个全连接层。
Xception简介
- 是一种深度卷积神经网络,由Google于2017年提出。Xception是由 “Extreme Inception” 衍生而来,表示对 Inception 架构进行极端的改进。
- Xception在Inception架构的基础上进行改进,引入深度可分离卷积来增强网络性能,将卷积操作拆分为深度卷积和逐点卷积两个步骤的结构,并在一定程度上减少了参数数量
- Xception基于Keras深度学习框架,大约有2100万个可训练参数,在ImageNet数据集上具有高性能。
- 输入图像均采用灰度化处理,并自动降采样到固定尺度:299 x 299像素。
- DeepSlice调用(在ImageNet数据集上的预训练模型)Xception进行微调。
DeepSlice模型参数:使用MSE均方误差损失函数和Adam优化器进行模型优化。初始学习率=0.001,batch_size=8。DeepSlice模型微调:初始冻结Xception的所有层,训练随机初始化的Dense层,然后迭代解冻,直到整个模型解冻。- 在 RTX 2080 Ti 工作站上
大约需要训练3~4天才能达到收敛。
全连接层由9个具有线性激活函数的输出神经元组成。通过分析冠状小鼠脑图像,并预测与图像的三个角坐标对应的输出向量
(Oxyz, Uxyz, Vxyz) = [ox, oy, oz, ux, uy, uz, vx, vy, vz]
数据集下载链接:使用来自艾伦脑科学研究所(Allen Institute for Brain Science)提供的冠状小鼠脑切片,该数据集已经与CCF对齐。
- 阶段一(效果较差):
IHC + FISH + Nissl + ISH + BDA + S2P
- 来自艾伦数据库的 131k 张切片
- 阶段二(增强训练):
S2P + Synthetic Nissl + Synthetic S2P
- 来自艾伦连接图谱的 443k 张切片
IHC: 免疫组织化学:用于检测组织中蛋白质表达的技术。通过使用抗体来标记特定的蛋白质,从而在组织切片中可视化这些蛋白质的分布。FISH: 荧光原位杂交:用于检测DNA序列的技术。通过使用荧光标记的探针来结合目标DNA,从而在细胞或组织中可视化特定基因的位置。Nissl: 尼氏染色:用于染色神经组织的技术,通常用于显示神经元和神经胶质细胞的分布和形态。ISH: 原位杂交:检测RNA序列的技术。类似于FISH,但通常用于检测特定基因的mRNA。BDA: 生物素-蛋白质-抗生素复合物追踪:用于神经追踪的技术,通过注射生物素标记的物质来标记和追踪神经通路。S2P: 拉曼光谱图像:用于获得样本中化学成分信息的技术,通过使用拉曼光谱技术来获得高分辨率的化学图像。Synthetic Nissl(合成尼氏染色): 通过模型或计算方法合成的类似于尼氏染色的图像,而不是通过实际的尼氏染色技术获得的图像。Synthetic S2P(合成拉曼光谱图像):通过模型或计算方法合成的拉曼光谱图像,而不是通过实际的拉曼光谱技术获得的图像。
1.3、优化策略
1.3.1、开发了一个基准数据集(GT)
主要原因:用于训练的数据缺乏 “地面真相” 的注册数据集。
(1)Allen组织学数据集很大,由不同染色技术获取。
(2)在阶段一图像中,除了S2P数据集具有高度精确配准,其余均容易出现撕裂、气泡和变形。
优化策略:
开发了一个基准数据集,由来自不同实验室的、使用常见染色技术处理的、7个小鼠大脑的305个切片组成。
使用 QuickNII 工具分别对7个脑进行手动注册:3名新手(本科生)、2名中级研究人员(研究生)、2名神经解剖学专家(具有十年经验)。- 模型训练:随机指定3个大脑作为
验证,4个大脑作为测试。
1.3.2、构建了阶段二的训练数据集(增强训练)
主要原因:在阶段一训练时,由于部分数据集的不对齐,导致模型的准确性较差。
优化策略:
(1)构建第二个训练数据集:S2P、合成Nissl、合成S2P。 Qslicer工具:用于合成数据集(Qslicer.zip=1.1GB)
(2)经验证,S2P配准精确高,因此分别在两个训练阶段都加入了S2P。
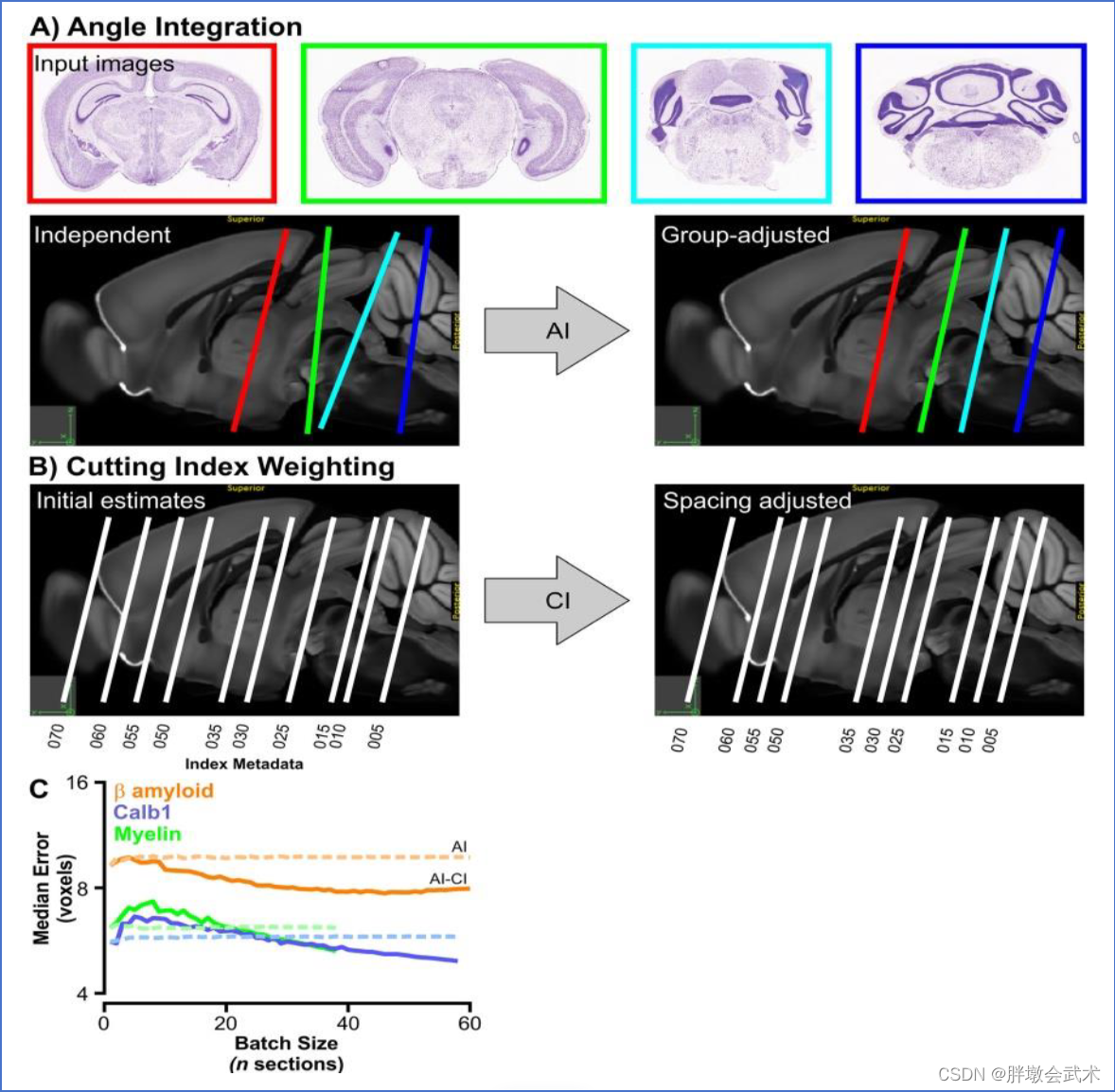
1.3.3、角度集成 + 切割索引(论文核心)
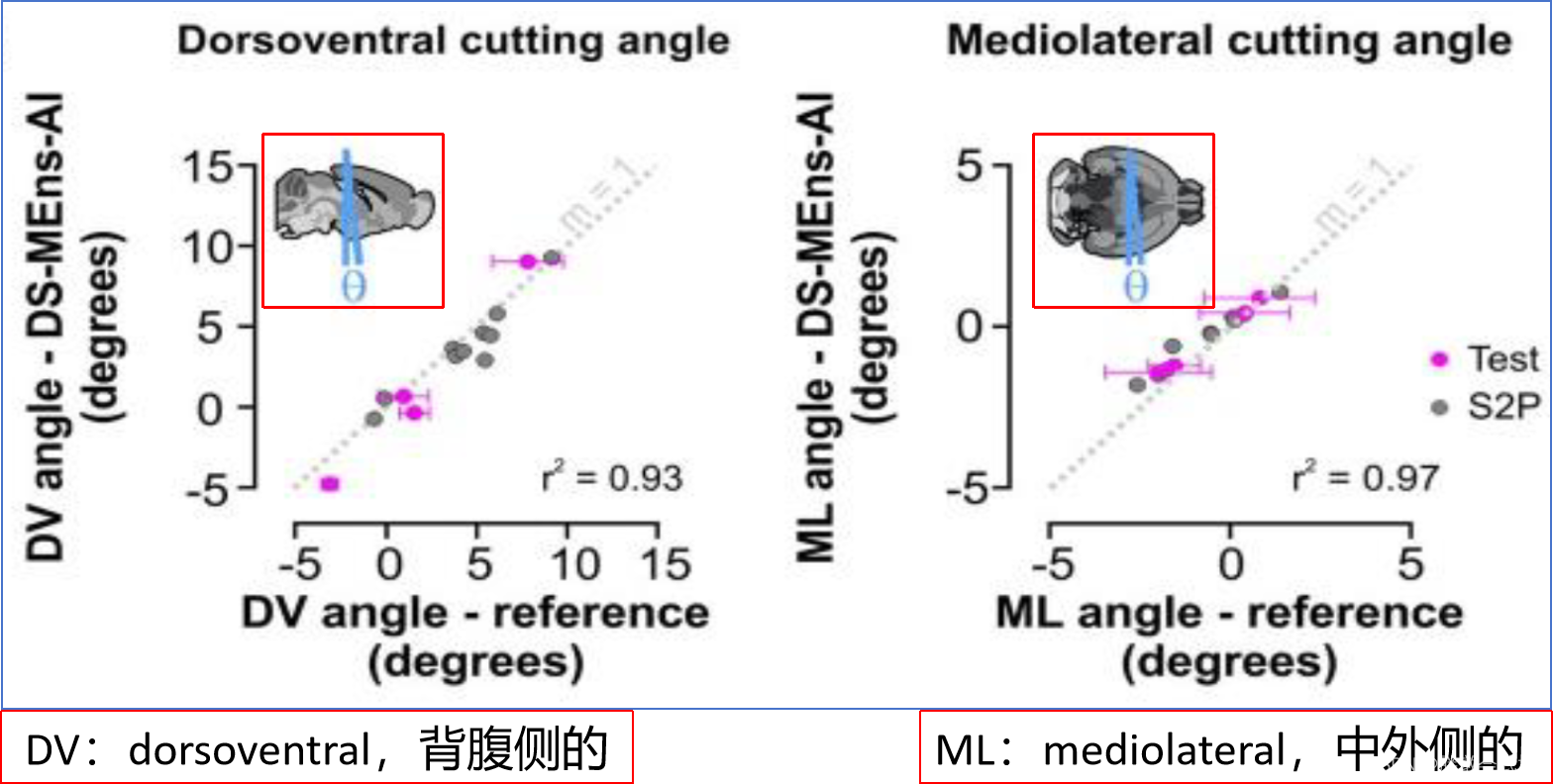
角度集成(Angle Integration,AI):由于连续切割的大脑切片时具有相同的切割角度的,因此需要将数据集中所有切片的(背腹侧和中外侧)的切割角度进行归一化,使其生成的角度等于预测的平均值。切割索引(cutting index metadata,CI):表示切片的切割顺序,在一个图像文件的元数据中,以文件名显示:slice1、slice2…。通过加权CI调整切片间的间距,提高了预测精度。
角度集成AI:所有组织切片都来源于冠状小鼠,但若在冠状面的连续切割中出现角度不一致,如AI左侧图中的线条表达的是矢状面的切割角度。切割索引CI:如CI左侧图中的线条表达的是矢状面的切割角度(均匀),但每个切割索引的位置不均为。如:060+055+050的间距是5,070+060的间距也是5,但是在图像中的位置都是5。
图形化理解:背腹侧 + 中外侧
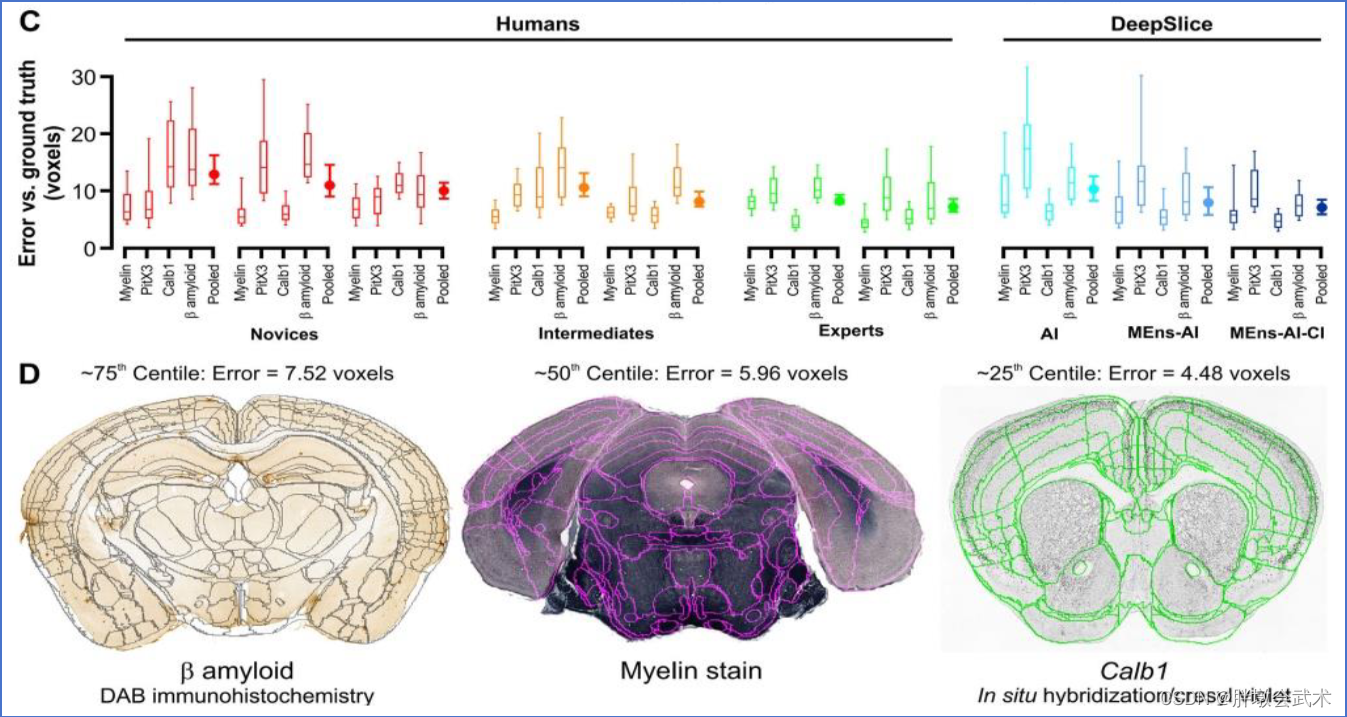
1.4、配准结果展示
整体的配准效果还是很不错的,但是在细节上的精度不佳。
1.4.1、必读信息
由于博主精通Elastic图像配准,可能是有一些先入为主的观念,故一直都不能理解:为什么DeepSlice模型只预测了图像的9个角坐标就可以实现图像配准???
经过对论文和源码的反复阅读,深入研究,终于领悟其中的要点。
总结如下:
- (总结1)论文中的全部数据集均与CCF对齐(训练+测试):这意味着输入图像与Atlas模板都是标准化图像,区别只在于输入图像与Atlas模板存在角度不对齐问题。 该操作是DeepSlice模型的必要条件
- (总结2)使用DeepSlice模型训练9个角坐标(用于矫正输入图像与Atlas模板的角度偏差),然后使用角坐标对Atlas进行
" 角度集成 "(目前只支持背腹侧与中外侧的角度矫正) 。即:将Atlas模板在9个角坐标的基础上进行反角度变换,使其与输入图像对齐。- (总结3)
" 加权 - 切割索引 ":根据输入图像的文件名称(切片编号),在Atlas模板中索引相匹配的帧图像。 该操作同样验证了总结1
- 加权表示只是作为辅助选项,训练模型具有自主能力。
- (实际操作的结果显示):将文件名 slice197 修改为 slice007,输出仍是与slice197对应的Atlas模板。
1.4.2、适用范围
- 训练数据全部来自艾伦脑科学研究所提供的均已经与CCF对齐的数据集,用于与Atlas对齐。
- 故对于非标准化图像的效果待评估(理论上不支持)
DeepSlice调用Xception在ImageNet数据集上的预训练模型进行微调。故输入图像均采用灰度化处理,并自动降采样到固定尺度 299 x 299 像素。
- 因此使用高分辨率图像并不会带来优势,反而会增加处理时间。
- 只适用于冠状面切片,未来将发布一个矢状面和横段面的切片组织学。
- 依赖于视觉线索来确定位置。亮度+对比度敏感,且对遮挡组织、畸形组织的表现不佳。
- 核心优势在于角度集成、加权 - 切割索引:对于单帧图像的配准较差。为了获取最佳效果,每批最少使用10-20个切片。
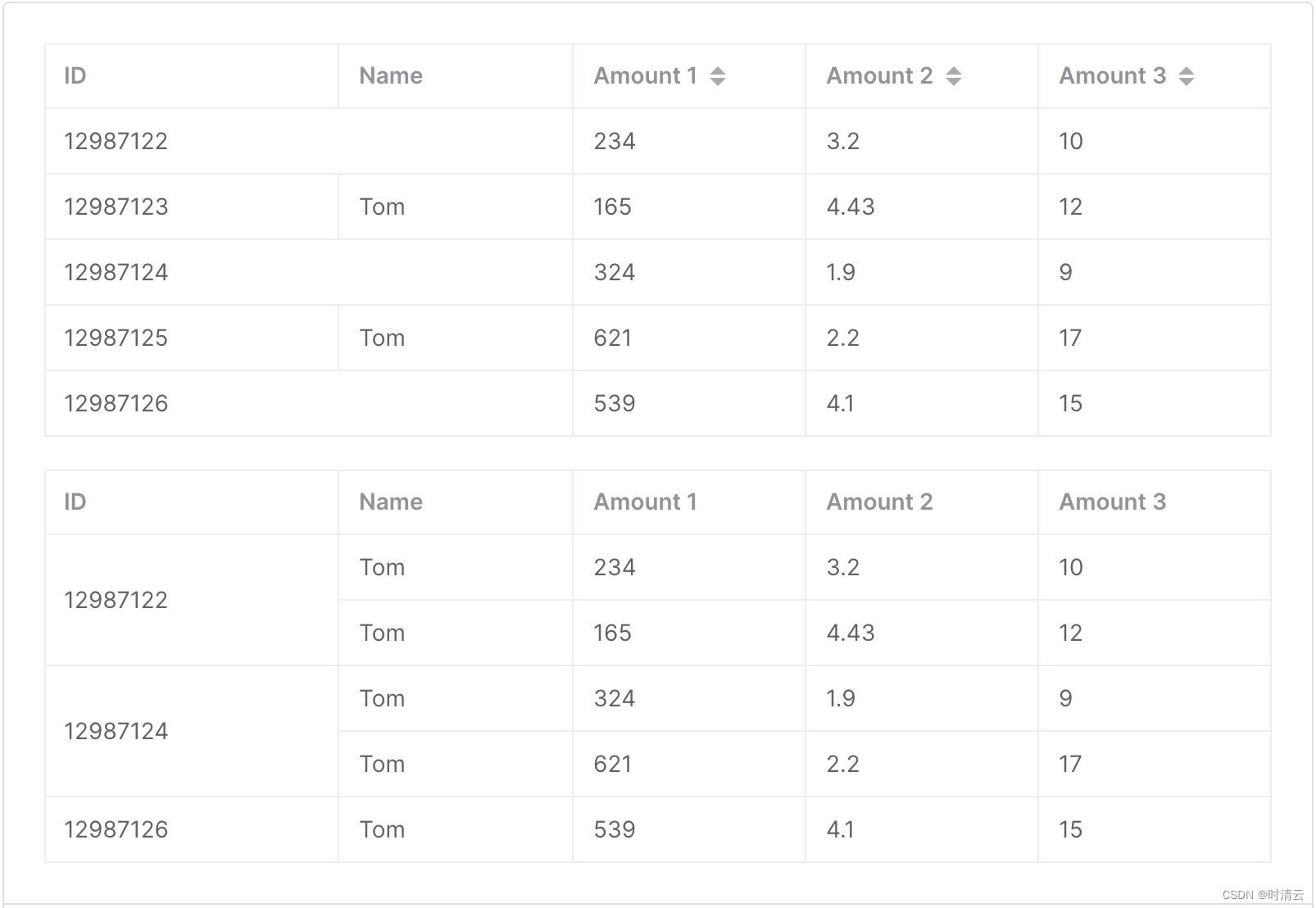
1.4.2、论文结论 + 个人看法
| 论文结论 | 个人看法 |
|---|---|
可靠地(只适用于冠状切片) |
整体的配准效果不错,但在细节上的精度不佳。 |
论文摘要:速度提高了1000倍以上(人工标记需要几个小时,而DeepSlice只需要秒级) |
只和人工标记比较,有些噱头。 |
与切片配准算法相比,能够泛化不同的成像模式和染色类型 |
阶段一 + 阶段二(虽然输入图像的类型较多,但除了S2P具有高精度,其余都很差) |
| 不需要图像预处理或编码能力 | 深度学习的天生优势 |
二、环境配置
2.1、无需安装的Web应用
若想使用 DeepSlice 但不想安装,请查看 DeepSlice Flask,这是一个功能齐全的 Web 应用程序,它允许您上传数据集并下载对齐的结果。Web 界面由Michael Pegios开发。
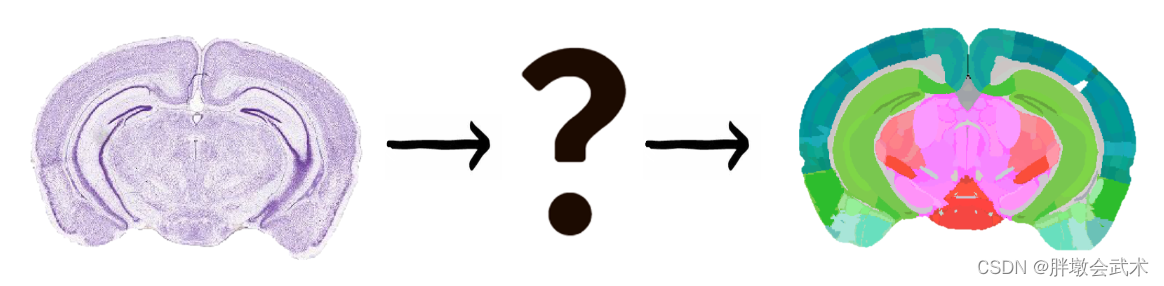
2.2、项目安装
2.2.1、创建虚拟环境
【Python虚拟环境】创建 + 激活 + 查看 + 退出 + 复制 + 删除
2.2.2、配置相关环境
【深度学习环境配置】Anaconda +Pycharm + CUDA +cuDNN + Pytorch + Opencv(资源已上传)
# Install the required packages
pip install numpy
pip install Python==3.7
pip install scikit-learn
pip install scikit-image
pip install tensorflow==1.15.0
pip install h5py==2.10.0
pip install typing
pip install pandas==1.3.5
pip install requests
pip install protobuf==3.20
pip install lxml
详细请看官网介绍:https://github.com/PolarBean/DeepSlice
通用知识
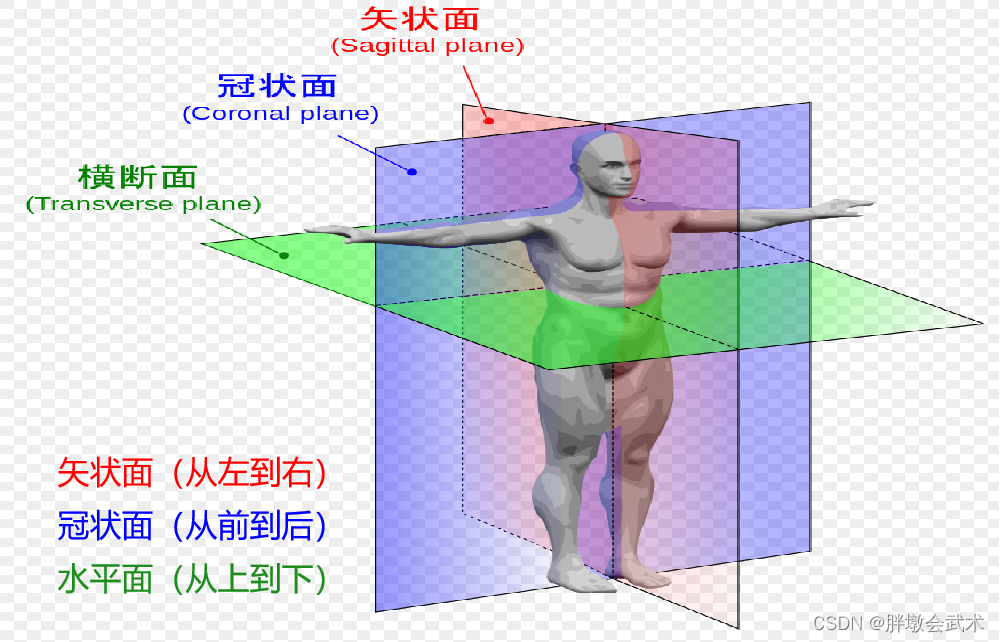
(1)CCF 和 Atlas
CCF 和 Allen 都是与神经科学和脑研究相关的工具,由艾伦脑科学研究所(Allen Institute for Brain Science)开发。
艾伦通用坐标框架(Allen Common Coordinate Framework,CCF):是一个标准化的三维空间坐标框架,用于定位、比较和分析小鼠不同脑区的解剖结构。
艾伦脑图谱(Allen Brain Atlas):是一个基于 CCF 的高分辨率三维脑图谱,用于理解脑区的解剖学信息、基因表达信息、功能区域的标定、神经元连接图等。
- CCF提供一个共同的坐标系统,使研究人员能够一致地定位脑区,以便进行数据比较和整合。
- Allen提供丰富的脑科学数据和工具,帮助研究人员深入探索小鼠脑的结构、功能以及基因表达等方面。
(2)冠状面 + 矢状面 + 横断面