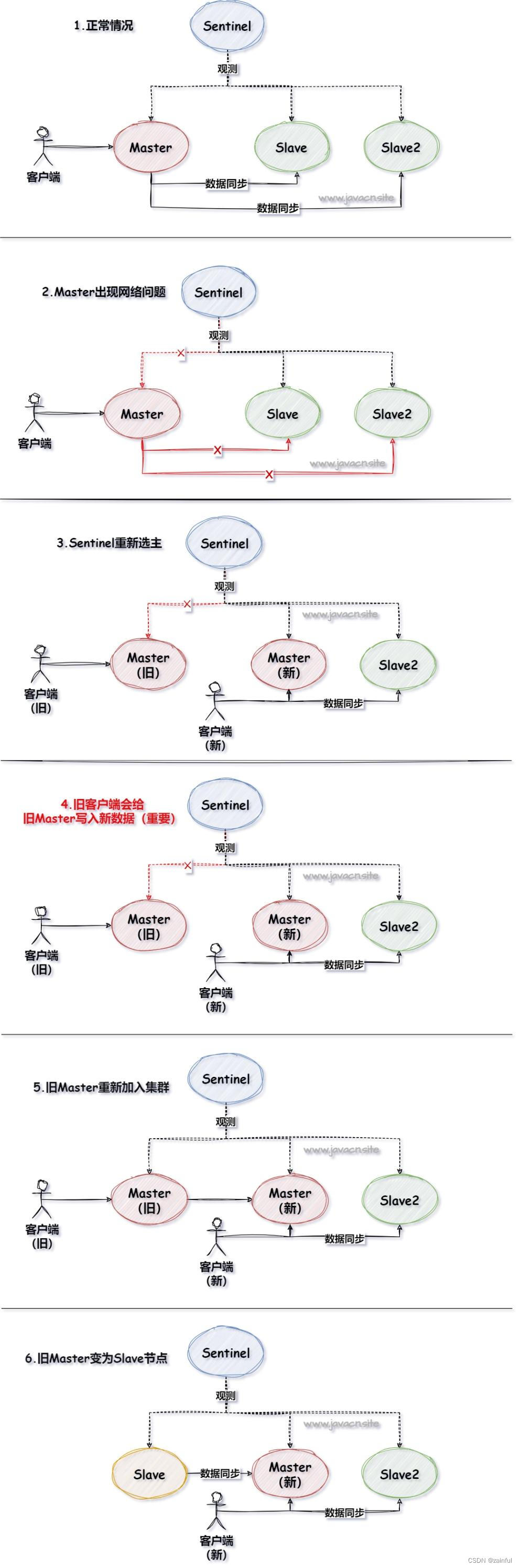
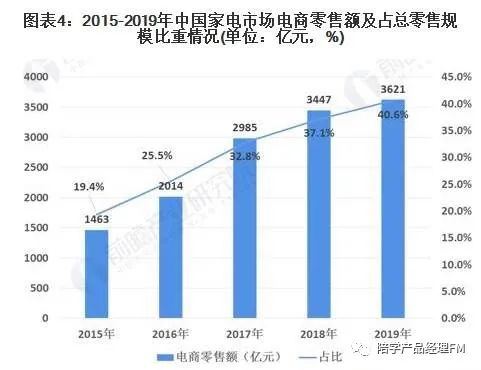
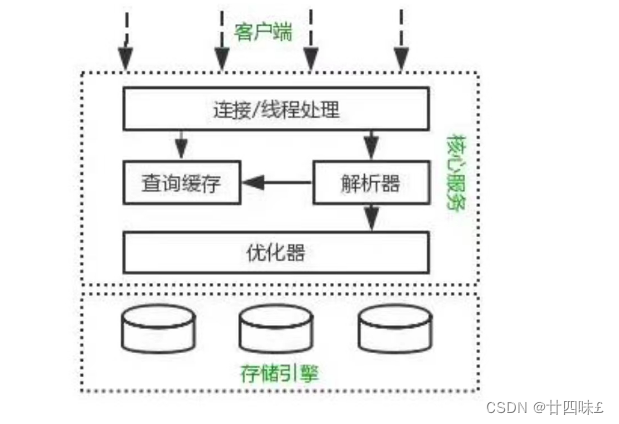
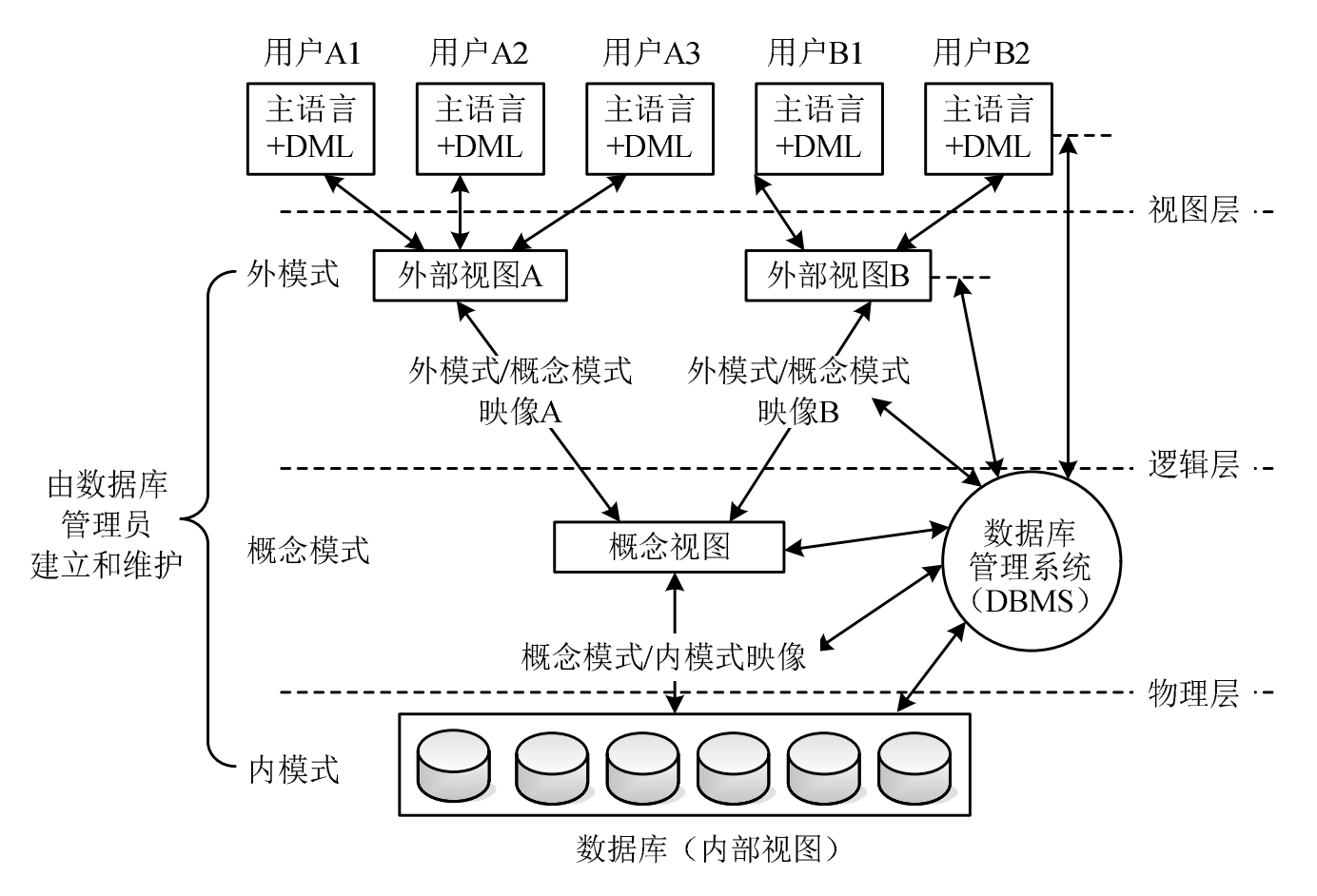
亚组分析、P交互、P趋势是什么?如何计算呢?
(1)亚组分析如何计算?
(2)P交互作用的计算方法?
(3)P趋势如何计算?
(1)亚组分析如何计算?
本篇不说理论,直接以R语言的lung数据集为例进行介绍。
除P交互的外,亚组分析和P趋势的计算以logistic模型为例进行说明:
自变量:sex(性别),age(年龄),ph.ecog(ECOG评分),wt.loss(过去六个月的体重减轻)
因变量:status(是否死亡)
交互作用我们主要关注性别和年龄的交互。
假设:我们主要关注性别和死亡的关系?
首先:由于要研究性别和死亡的关系,因此我们建立的主要分析模型如下:
Logit(p)=β1sex+β2age+β3ph.ecog+β4wt.loss+残差
在这里,β1是我们关注的主要解释参数。age+ph.ecog+wt.loss都是协变量
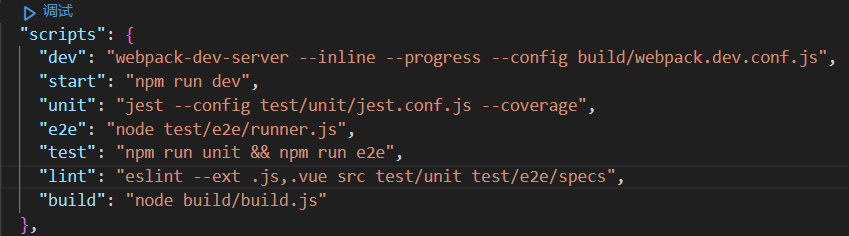
R语言代码:
#数据处理
library(rms)
library(dplyr)
data<-lung
#生成二分类新变量
data$age1 <- ifelse(data$age > 60,
c("1"), c("2"))
data$status<-data$status-1
str(data)#查看数据类型
#转化因子
a<-c("sex","age1")#填入需要转化的变量
data[,a]<-lapply(data[,a],as.factor)#转因子
#建立模型
#logistic模型:sex和status的关系
fit1 <- glm(status~sex+age1+ph.ecog+wt.loss,data=data,family = "binomial")
broom::tidy(fit1)
#自编函数,计算OR值
formatFit<-function(fit){
#取P值
p<-summary(fit)$coefficients[,4]
#wald值
wald<-summary(fit)$coefficients[,3]^2
#B值
valueB<-coef(fit)
#OR值
valueOR<-exp(coef(fit))
#OR值得95%CI
confitOR<-exp(confint(fit))
data.frame(
B=round(valueB,3),
Wald=round(wald,3),
OR_with_CI=paste(round(valueOR,3),"(",
round(confitOR[,1],3),"~",round(confitOR[,2],3),")",sep=""),
P=format.pval(p,digits = 3,eps=0.001)
)
}
formatFit(fit1)#得出OR值
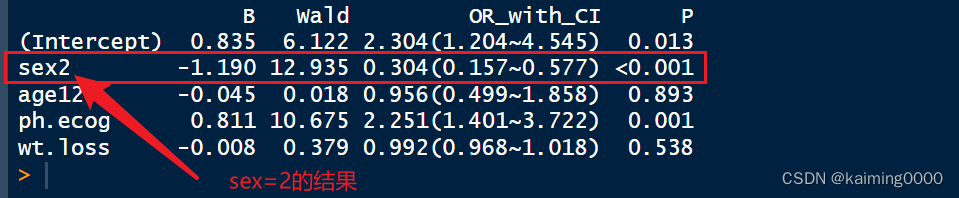
结果解释:OR=0.304,因此sex=2的死亡风险更低。
- 亚组分析如何计算
那么,为了进一步探讨,不同年龄组的性别和死亡的关系,我们需要将人群按照年龄组分为60岁以下和60岁以上两组,分别建立两个模型:
60岁以下:Logit(p)=β1sex+β3ph.ecog+β4wt.loss+残差
60岁以上:Logit(p)=β1sex+β3ph.ecog+β4wt.loss+残差
这时候模型中就去掉了age这个变量,然后会得到两个β1,这其实就是亚组分析,也叫分层分析。
R语言代码:
#亚组分析
data1<-subset(data,age>60)
data2<-subset(data,age<=60)
#60岁以上
fit2 <- glm(status~sex+ph.ecog+wt.loss,data=data1,family = "binomial")
#60岁以下
fit3 <- glm(status~sex+ph.ecog+wt.loss,data=data2,family = "binomial")
formatFit(fit2)
formatFit(fit3)

结果解释:OR=0.284和0.309,因此60岁以上和60岁以下,sex=2的死亡风险均更低。
(2)P交互作用的计算方法?
那么OR=0.284和0.309,两者相等吗?这时候就需要计算P交互值了。
P交互值的计算方法有两种,一种是传统方法,一种是似然比检验。
- 传统方法:在模型中加入主要解释变量和亚组变量的交互项,交互项的P值即为P交互,本例的模型如下:
Logit(p)=β1sex+β2age+β3sex*age+β4ph.ecog+β5wt.loss+残差
β3的P值即为P交互。
- 似然比检验:步骤有三步
步骤1:建立有交互的模型,即Logit(p)=β1sex+β2age+β3sex*age+β4ph.ecog+β5wt.loss+残差
步骤2:建立无交互的模型,即Logit(p)=β1sex+β2age+β3ph.ecog+β4wt.loss+残差
步骤3:比较这个模型差异,计算的P值即为P交互
若P交互<0.05,则说明OR=0.284和0.309在统计学上有差异,否则无差异。
下面分别给出线性模型、logistic模型、cox模型传统方法和似然比检验计算交互P的代码:
#模型1:线性模型
#有交互模型
fit1 <- glm(meal.cal~sex*age1+sex+age1+ph.ecog+wt.loss,data=data,family = "gaussian")
broom::tidy(fit1)
#无交互模型
fit2 <- glm(meal.cal~sex+age1+ph.ecog+wt.loss,data=data,family = "gaussian")
broom::tidy(fit2)
#似然比检验
anova(fit1,fit2,test="Chisq")
#模型2:logistic模型
# 有交互模型
fit1 <- glm(status~sex*age1+sex+age1+ph.ecog+wt.loss,data=data,family = "binomial")
broom::tidy(fit1)
#无交互模型
fit2 <- glm(status~sex+age1+ph.ecog+wt.loss,data=data,family = "binomial")
broom::tidy(fit2)
#似然比检验
anova(fit1,fit2,test="Chisq")
#模型3:Cox模型
# 有交互模型
fit1 <- coxph(Surv(time,status)~sex*age1+sex+age1+ph.ecog+wt.loss,data=data)
broom::tidy(fit1)
#无交互模型
fit2 <- coxph(Surv(time,status)~sex+age1+ph.ecog+wt.loss,data=data)
broom::tidy(fit2)
#似然比检验
anova(fit1,fit2,test="Chisq")
(3)P趋势如何计算?
由于P趋势的计算要求自变量至少为三种,因此,这里研究目的改为年龄和死亡的关系。
首先将年龄划分为<50,50~60,60~70,70及以上四组。
然后首先将年龄设置为哑变量,建立以下模型:
Logit(p)=β1age12+β2age13+β3age14+β4sex++β5ph.ecog+β6wt.loss+残差
我们会得到β1、β2、β3三个参数估计值,然后进一步得出OR1、OR2、OR3.
R语言代码:
###########P趋势的计算
#年龄分组
data <- within(data,{
age1[age<50]=1
age1[age>=50 & age < 60] = 2
age1[age>=60 & age < 70] = 3
age1[age>= 70] = 4
})
#变因子
data$age1<-as.factor(data$age1)
#设参照
data$age1<-relevel(data$age1, ref="1")
#建立模型
fit1 <- glm(status~age1+sex+ph.ecog+wt.loss,data=data,family = "binomial")
broom::tidy(fit1)
formatFit(fit1)
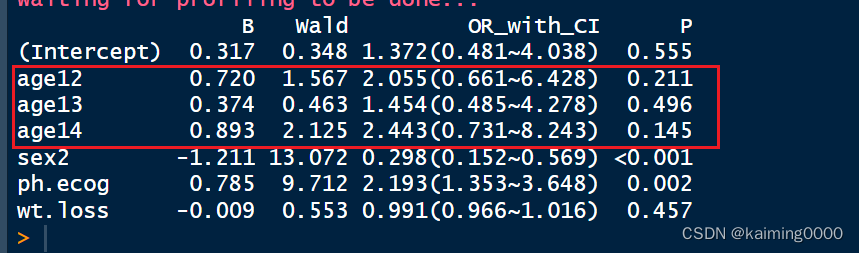
OR分别为2.055、1.454、2.433,这三个值在统计学有没有趋势性变化呢?这时候就需要计算P趋势值了。
目前有两种方法计算P趋势,以本例为例子进行说明
(1)将age变量编码为1、2、3、4,作为等级变量纳入模型,计算出的P值就是P趋势,P趋势的方向是正向还是反向,可以看估计的β值或OR值
(2)先将age变量编码为1、2、3、4,然后计算每一组的中位数,本例计算的中位数为。。。。,因此将原来的1、2、3、4替换成。。。。/,再作为等级变量纳入模型,计算出的P值就是P趋势,P趋势的方向是正向还是反向,可以看估计的β值或OR值
R语言代码:
############方法1:age1变为等级变量
#age1变为数值
data$age1<-as.numeric(data$age1)
#建立模型
fit1 <- glm(status~age1+sex+ph.ecog+wt.loss,data=data,family = "binomial")
broom::tidy(fit1)
formatFit(fit1)
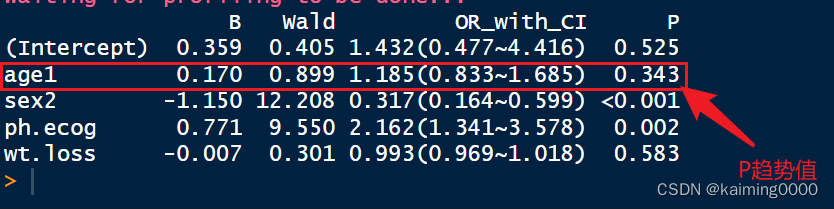
############方法2:age1各组的中位数变为等级变量
library(dplyr)
# median of groups
summarise(group_by(data,age1),
min(age),
max(age),
mean(age),
sd(age),
median(age),
quantile(age,0.25),
quantile(age,0.75),
)
#age的中位数:44,56,64,7
data$age2[data$age1 == 1]<- 44
data$age2[data$age1 == 2]<- 56
data$age2[data$age1 == 3]<- 64
data$age2[data$age1 == 4]<- 73
#建立模型
fit1 <- glm(status~age2+sex+ph.ecog+wt.loss,data=data,family = "binomial")
broom::tidy(fit1)
formatFit(fit1)
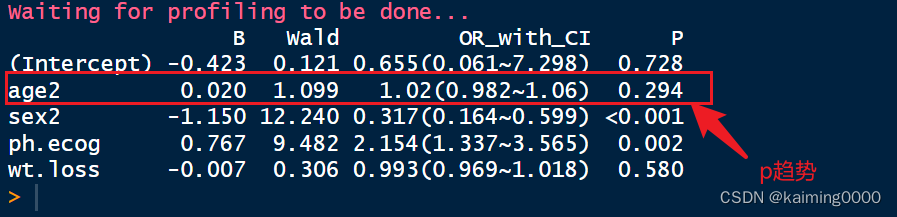
结果解释:P趋势值>0.05,因此不同年龄的死亡风险无趋势性变化。
这里可以发现两种做法结果不太一致,目前主流的做法基本是第2种做法,因此,小编建议大家参考第2种做法,当然第1种做法也没错。




![洛谷 <span style='color:red;'>P</span>1980 [NOIP2013 普及<span style='color:red;'>组</span>] <span style='color:red;'>计数</span>问题](https://img-blog.csdnimg.cn/direct/af6866831fb64b35bbec35ccc71b5242.gif)