概要
不常见的物种如何进行富集分析??
整体流程
提示:
step1 百度搜索注释物种–拉丁文名称
提示:拉丁文名要准确
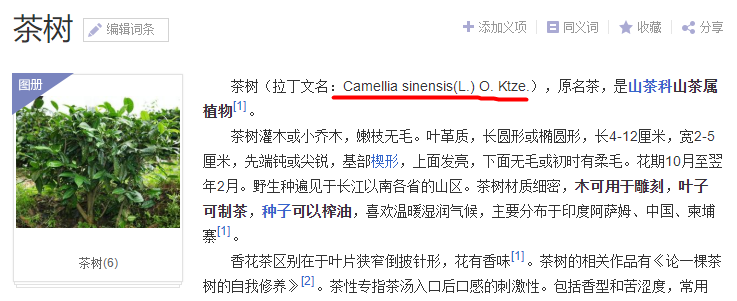
例如:[百度茶树](https://baike.sogou.com/v87233.htm?ch=frombaikevr&fromTitle=%E8%8C%B6%E6%A0%91)
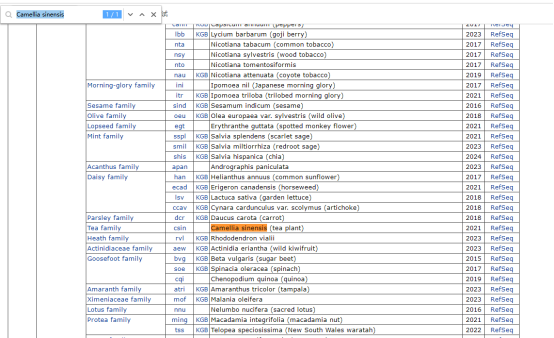
step2 注释官网搜索–该物种对应库–编号
官网:https://www.genome.jp/kegg/tables/br08606.html
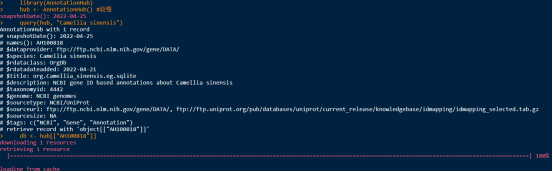
Ctrl +f : 页面搜索Camellia sinensis
library(clusterProfiler) library(AnnotationHub) hub <- AnnotationHub() #较慢 query(hub, "Camellia sinensis") db <- hub[["AH100818"]] organism <- "csin" #看官网有物种简称