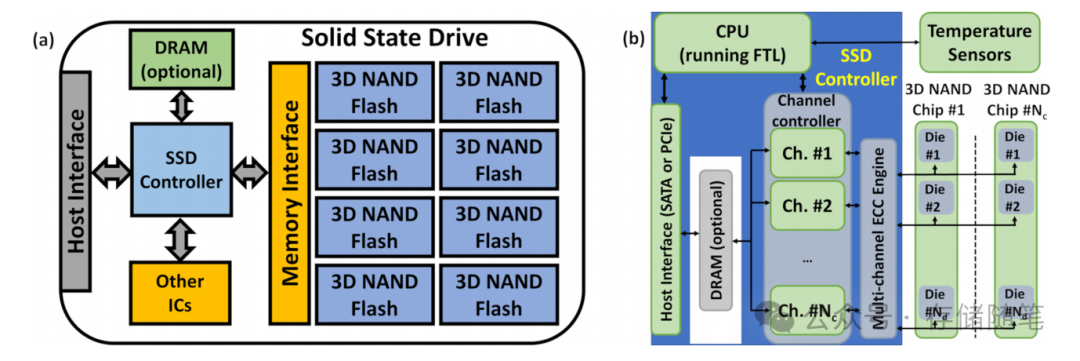
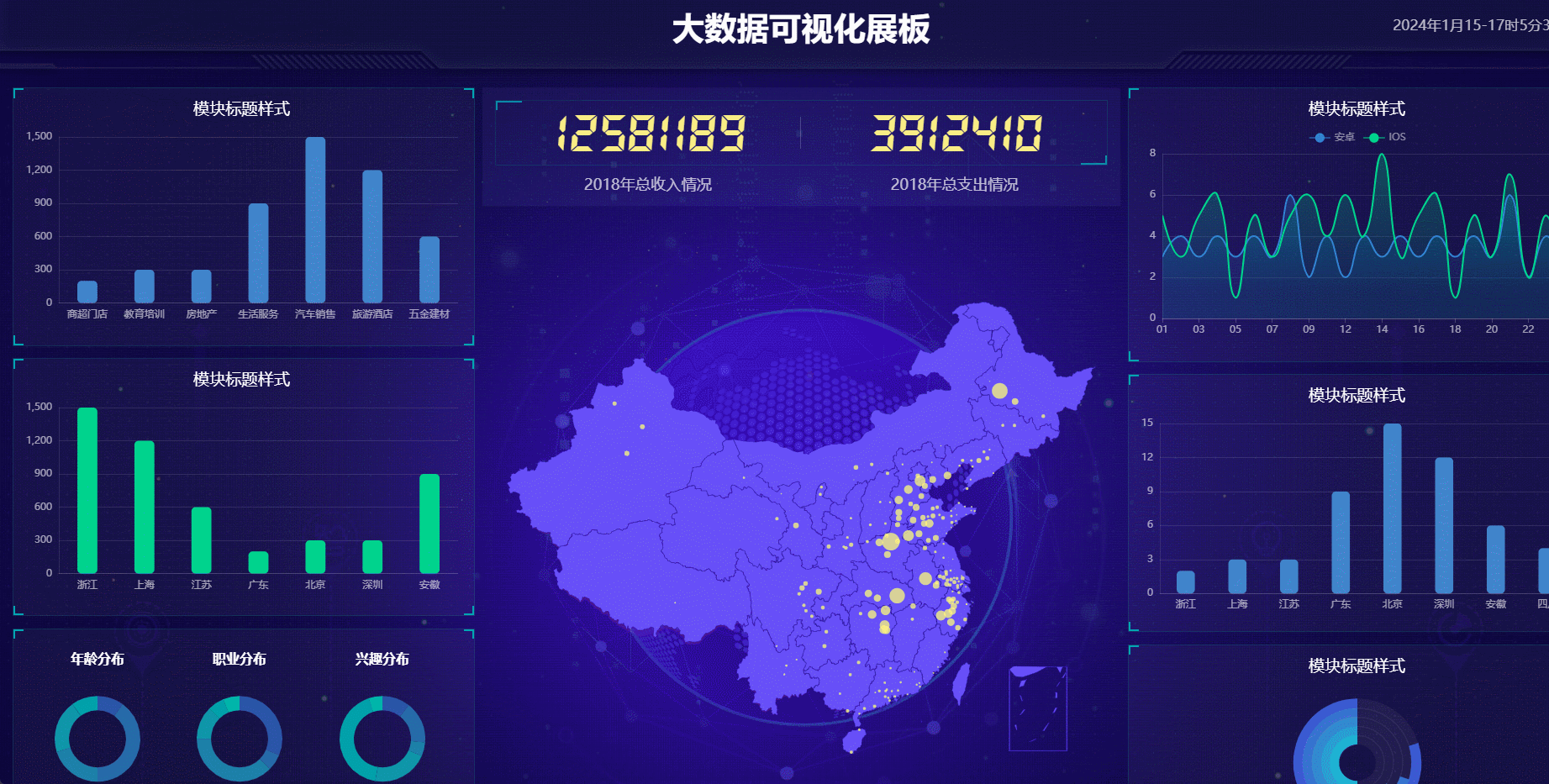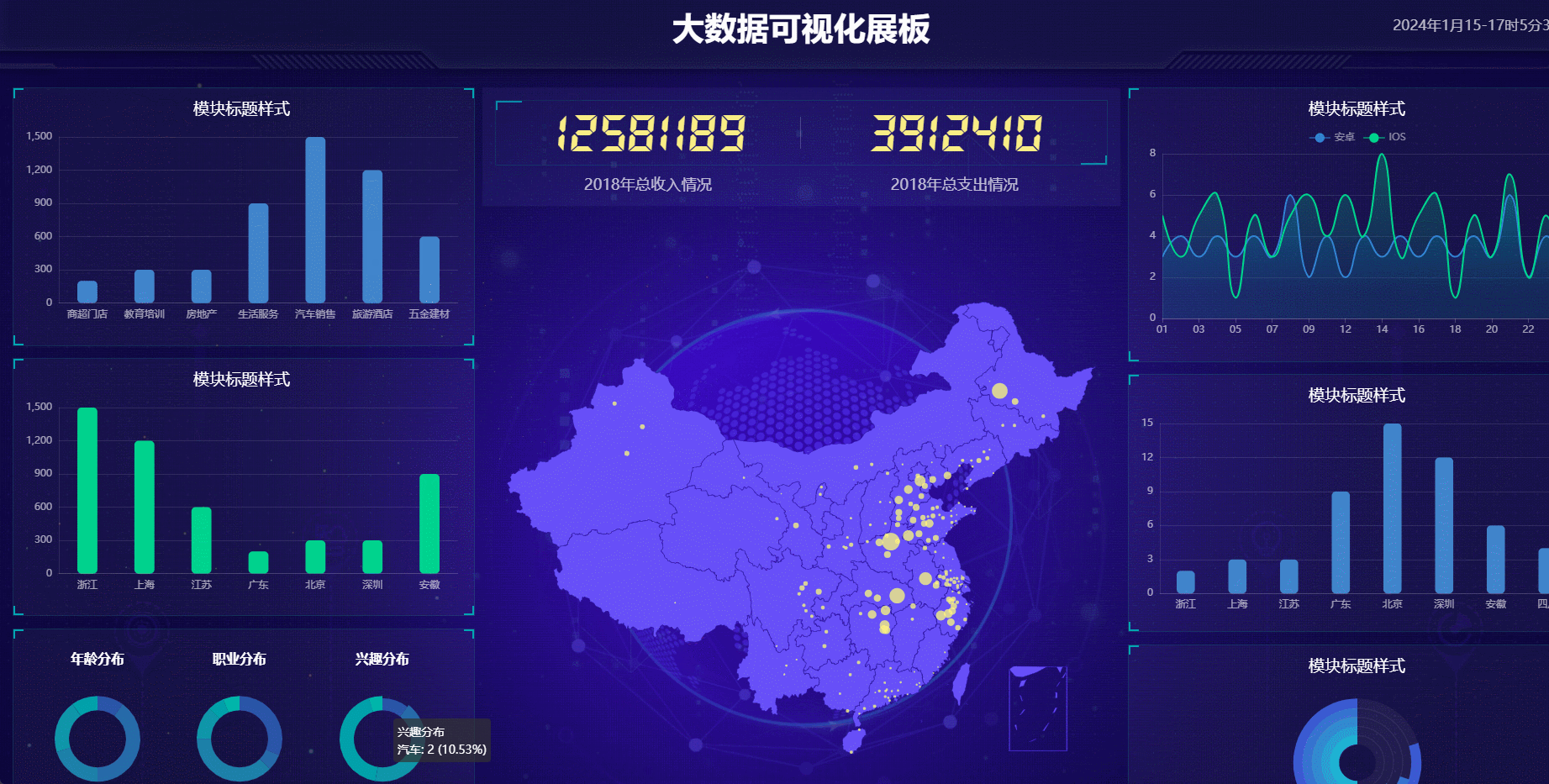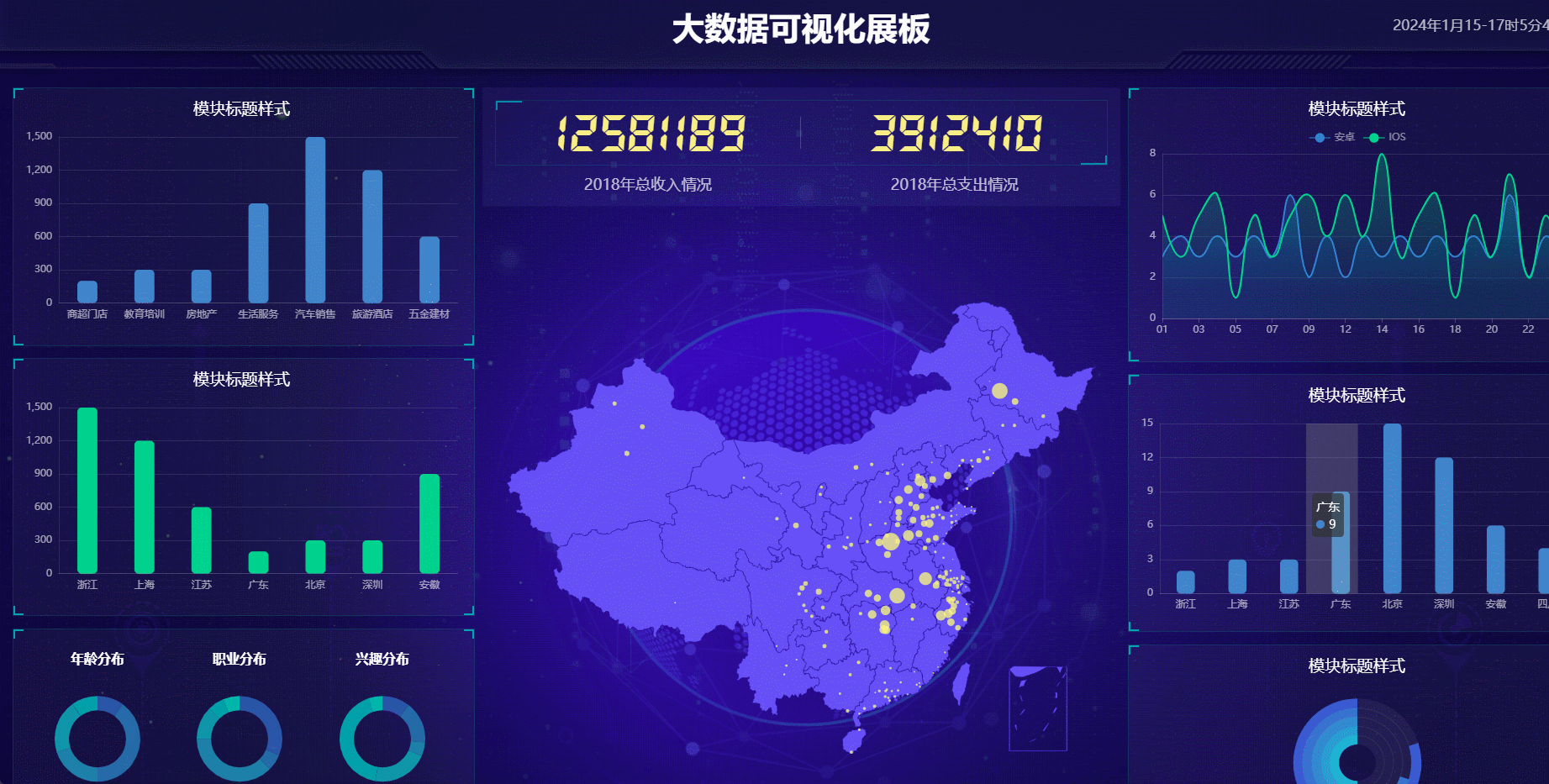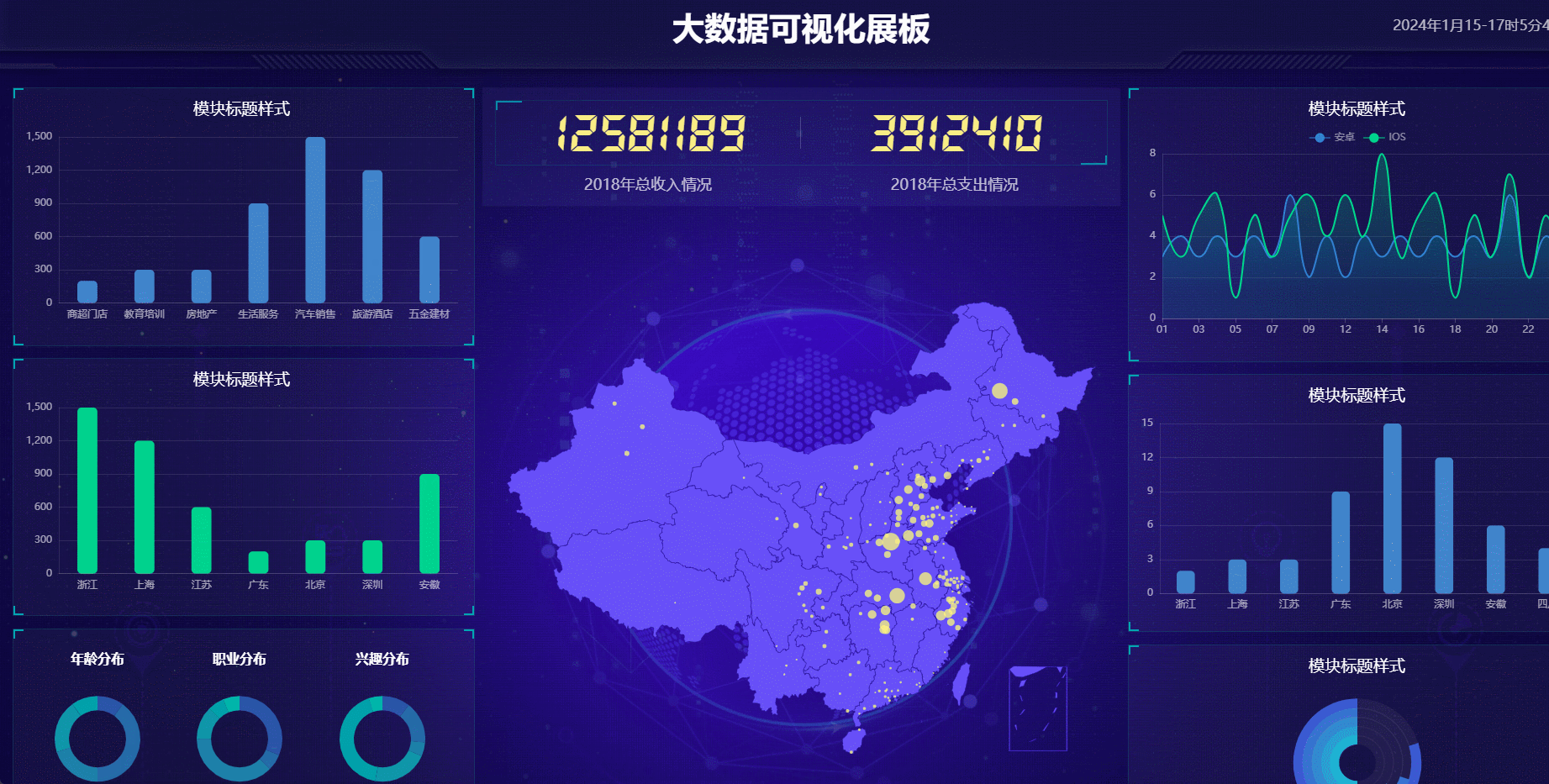
经常分析单细胞数据的同学都知道,10x Genomics 的细胞基因矩阵通常存储在三个关键文件中,这些文件共同描述了单细胞测序数据。这三个文件分别是:
Barcodes.tsv,通常是一列多行,每一行代表一个细胞。
Features.tsv,通常是两列多行,每一行代表一个基因,每行第一个是基因ID,第二个是对应的基因名称。
Features.tsv。通常是三列多行,前两行可以理解为标题。从第三行开始,第一个数n1代表基因(即Features.tsv中第n1行对应的基因),第二个数代表细胞(即Barcodes.tsv中第n2行对应的细胞),第三列是表达量。
通常来说,将这三个文件放在一个文件夹,并且命名正确,就可以直接使用Read10X函数来创建细胞矩阵了。
有的组由于原始数据处理的代码写的不够优雅,搞出的细胞-基因矩阵是tsv格式的也就算了,矩阵的行列都搞反了,搞成了每一列代表一个基因,每一行代表一个细胞,与seurat的RNA矩阵刚好相反。
如果你遇到的矩阵也是以列代表基因,行代表细胞,那么为了将完整矩阵改成10xGenomics的稀疏矩阵格式,你可以采用使用以下R代码将它改成符合10x genomics标准的稀疏矩阵三文件:
library(Matrix)
setwd("修改为输出文件夹路径")
mat1 <- read.table("修改为细胞-基因名矩阵文件,包含文件路径")
mat2 <- read.table("修改为细胞-基因ID矩阵文件,包含文件路径")
mat1 <- as.matrix(mat1)
mat2 <- as.matrix(mat2)
genename <- data.frame(Column1 = colnames(mat2),Column2 = colnames(mat1))
write.table(rownames(mat2), file = 'barcodes.tsv', sep = '\t', row.names = FALSE, col.names = FALSE, quote=F)
write.table(genename, file = "genes.tsv", sep = "\t", row.names = FALSE, col.names = FALSE, quote=F)
mtx1<-Matrix(t(mat1))
writeMM(obj = mtx1, file='matrix.mtx')